10x Genomics 在分析軟件這一塊還是非常給力的,開發的每個軟件都非常好用而且操作簡單,特別是安裝方便,基本解壓后就能用。對于 10x 的空間轉錄組數據主要用 10x 自己開發的 Space Ranger 來進行前期的處理。
軟件下載地址
https://support.10xgenomics.com/spatial-gene-expression/software/downloads/latest
可以直接點擊下載,也可以用下面的 curl 或 wget 命令下載。
基因組下載
在前面同一個頁面往下拉就能看到。
10x 提供了專門的用于空間轉錄組分析的基因組,目前較新的基因組版本是 2020 年 6 月更新的,基因注釋為 Ensembl 98 版本,主要包括人和小鼠。
系統要求
Linux 系統上運行底限要求:
8 核 Intel 或 AMD 處理器(建議使用 32 核)
64GB RAM(建議 128GB)
1TB 可用磁盤空間
64 位 CentOS / RedHat 6.0 或 Ubuntu 12.04
集群模式下運行需要額外滿足以下要求:
每個節點 8 核 Intel 或 AMD 處理器
每個內核 6GB RAM
共享文件系統(例如 NFS)
SGE 或 LSF 批處理系統
軟件安裝
1,解壓 SpaceRanger 文件
1 tar -xzvf spaceranger-1.1.0.tar.gz
2,解壓參考基因組文件
1 tar -xzvf refdata-gex-GRCh38-2020-A.tar.gz
3,加入環境變量
1 export PATH=/opt/spaceranger-1.1.0:$PATH
輸入文件準備
Fastq 序列文件
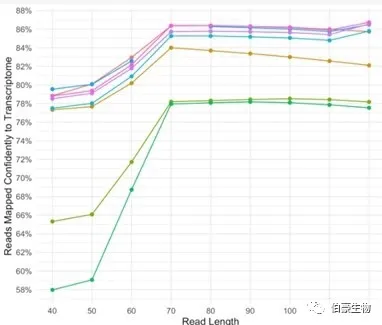
10x 建議測序 read2 設置 90 個堿基的長度 。 從下面 10x 測試的 6 個樣本來看,reads 序列長度達到 70bp 以后,mapping 率就不太變化了,有些樣本在長度大于 90bp 以后 mapping 率反而下降了。10x 給出這樣的建議是考慮到建庫后插入片段太短的話,太長的序列里面會包含部分 polyA 甚至 UMI 序列反而影響比對效果。 因為國內目前基本上是測 2x150bp 的,如果建庫片段過短比對率較低的話建議將 read2 剪切成 90bp 來分析效果應該會好一點。
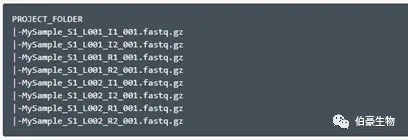
Fastq 文件名建議是如下格式的,以免運行報錯。
圖像文件:
Space Ranger 支持兩種樣式的顯微鏡圖像:用蘇木精和曙紅(H&E)染色的明場圖像,必須是 24 位彩色 TIFF,16 位灰度 TIFF 或 JPEG。Space Ranger 要求任一方向上的圖像至少為 2000 像素。
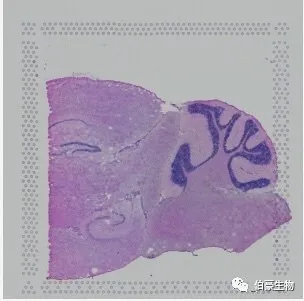
注意:圖像邊框的四個角位置是固定的,比如說 三角形在左下角 ,如果掃描出來的圖片四個角的位置變了需要先手動調整過來再使用。
軟件運行
一般我們拿到的都是直接是 fastq 序列文件了,所以可以直接跑 spaceranger count,不需要再去運行 spaceranger mkfastq 了。
spaceranger count 運行時圖片對其有兩種方式,一種軟件自動識別圖片進行對齊,另外一種就是先用 Loupe 軟件手動對齊,生成對于 json 文件提供給后面的軟件,后期會介紹怎么來手動調整圖片。
自動對齊
1 $ cd /home/jdoe/runs
2 $ spaceranger count --id=sample345 \
3 --transcriptome=/opt/refdata/GRCh38-3.0.0 \
4 --fastqs=/home/jdoe/runs/HAWT7ADXX/outs/fastq_path \
5 --sample=mysample \
6 --image=/home/jdoe/runs/images/sample345.tif \ 7
7 --slide=V19J01-123 \
8 --area=A1
手動對齊
1 $ cd /home/jdoe/runs
2 $ spaceranger count --id=sample345 \
3 --transcriptome=/opt/refdata/GRCh38-3.0.0 \
4 --fastqs=/home/jdoe/runs/HAWT7ADXX/outs/fastq_path \
5 --sample=mysample \
6 --image=/home/jdoe/runs/images/sample345.tif \
7 --slide=V19J01-123 \
8 --area=A1 \
9 --loupe-alignment=sample345.json
參數說明:
--Id:結果輸出文件夾名稱
--Transcriptome:基因組目錄
--Fastqs:fastq 文件木蘭路
--Sample:原始樣本名
--Image:鏡像圖片文件
--Slide:使用的 10x 芯片型號
--area:樣本所在芯片的區域(四通道芯片位置從上到下分別為 A1、B1、C1、D1)
--loupe-alignmen: 圖片手動對齊生成的 json 文件,如果是自動對齊不要需要此參數
另外,spaceranger 默認使用系統上可用的所有內核來執行管。可以使用 --localcores 選項來限制使用內核的數量。同理,可以使用 -localmem 來限制使用內存的大小。
結果輸出:
成功運行后會到的下面的信息(包括主要的輸出文件)
1 2016-11-10 16:10:09 [runtime] (join_complete) ID.sample345.SPATIAL_RNA_COUNTER_CS.SPATIAL_RNA_COUNTER_CS.SUMMARIZE_REPORTS 2
2
3 Outputs:
4 - Run summary HTML: /opt/sample345/outs/web_summary.html
5 - Outputs of spatial pipeline: /opt/sample345/outs/spatial
6 - Run summary CSV: /opt/sample345/outs/metrics_summary.csv
7 - BAM: /opt/sample345/outs/possorted_genome_bam.bam
8 - BAM index: /opt/sample345/outs/possorted_genome_bam.bam.bai
9 - Filtered feature-barcode matrices MEX: /opt/sample345/outs/filtered_feature_bc_matrix
10 - Filtered feature-barcode matrices HDF5: /opt/sample345/outs/filtered_feature_bc_matrix.h5
11 - Unfiltered feature-barcode matrices MEX: /opt/sample345/outs/raw_feature_bc_matrix
12 - Unfiltered feature-barcode matrices HDF5: /opt/sample345/outs/raw_feature_bc_matrix.h5
13 - Secondary analysis output CSV: /opt/sample345/outs/analysis
14 - Per-molecule read information: /opt/sample345/outs/molecule_info.h5
15 - Loupe Browser file: /opt/sample345/outs/cloupe.cloupe
16
17 Pipestance completed successfully!
空間轉錄組測序服務優勢
高質量切片: 切片、貼片經驗豐富,針對不同組織優化了解決方案。
流程化分析: 完善的分析流程,準確快速解析空間轉錄組數據。
標準化內控: 豐富的實操經驗構建了標準化的內控體系。
專業化團隊: 資深的技術團隊具有多年項目方案設計、實驗操作、售后分析等經驗。
全流程服務: 提供組織冷凍包埋、貼片、切片、透化、建庫測序及數據分析的全套服務。
更多伯豪生物人工服務:


