 |  |

基因表達具有時間和空間的特異性,通過對不同時間點的樣本取材,使用單細胞轉錄組測序技術能夠解析時間維度上細胞類型和基因表達的變化過程。然而單細胞測序實驗的前提是組織必須通過機械分離或酶解消化成單細胞懸液,此過程不可避免的丟失了組織中細胞所處的原始位置信息,也導致了細胞間的通訊網絡被打破,這使我們難以獲得組織中不同區域的細胞構成和基因表達狀態,以及不同功能區之間的基因差異表達等信息。現有的原位表達圖譜主要是通過報告基因或原位雜交等技術來實現,但是這些方法實現比較困難,并且通量低,限制了多樣本、高時效的應用。而空間轉錄組技術則可以高 效的檢測組織中空間原始位置上的基因表達模式。

▲圖 空間轉錄組被評為 2020 年度技術
注:圖片來源互聯網 - 侵刪
空間轉錄組(Spatial Transcriptomics)就是將基因的表達情況與關注的組織切片的免疫化學染色圖像進行整合,從而將組織內不同細胞的基因表達信息定位到組織的原始空間位置上去,進而直接觀測組織中不同部位功能區基因表達的差異。空間轉錄組技術利用了常規的原位技術和組學技術兩方面的優勢。實際上空間轉錄組已不是新名詞,2016 年 Joakim Lundeberg 的 Spatial Transcriptomics 技術在 Science 上發表,2017 年景乃禾老師的 GEO-seq 技術在 Nature Protocols 上發表 [2]。目前已發表的關于空間轉錄組技術有主要有 Spatial Transcriptomics, Slide-seq, LCM-seq, seqFISH, MERFISH, Liver single cell zonation, Geo-seq 和 Tomo-seq, 涉及物種有人、小鼠、果蠅、秀麗隱桿線蟲、斑馬魚、擬南芥、楊樹和云杉等。
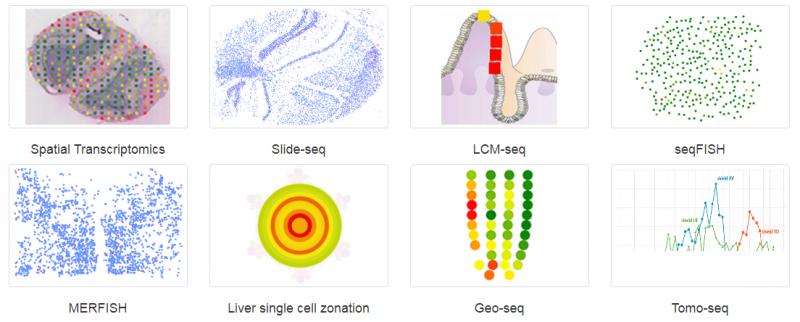
▲圖 常見空間轉錄組技術
注:圖片來源互聯網 - 侵刪

其中,瑞典皇家理工學院的 Joakim Lundeberg,基于芯片和空間條形碼技術發明了高通量的空間轉錄組測序方法,并創建了 Spatial Transcriptomics 公司。2018 年底 10X Genomics 宣布收購 Spatial Transcriptomics,并于 2019 年發布 Visium 空間基因表達解決方案(Visium Spatial Gene Expression Solution)。10X Genomics 公司提供的 Visium 空間基因表達解決方案是高通量空間轉錄組的商業化解決方案,其可以檢測完整組織切片的總 mRNA,將總 mRNA 的空間信息與形態學內容相結合,并繪制所有基因表達發生的位置,獲得完整的基因表達圖譜;在確定不同細胞群的同時保留空間位置,為細胞功能、表型和組織微環境中位置的關系提供了重要信息。

空間轉錄組學技術優勢:是轉錄組學研究領域的新方向,也是研究細胞異質性方面的新方法。
(1)準確定位:探針有效定位組織中 RNA 空間位置,實時了解組織中的轉錄組天然狀態。
(2)簡單易行:芯片設計簡單易操作、重復性高、可快速獲得高分辨率空間轉錄組信息。
(3)適用性廣:有效應用于發育生物學、腫瘤生物學、腦神經科學、植物研究等各生物學領域。
1、伯豪個性化方案:空間轉錄組測序研究需求千差萬別,伯豪生物專業科研團隊針對客戶需求一對一項目建議,為客戶定制空間基因表達解決方案。
2、組織保存液:空間轉錄組技術對樣本質量要求高,新鮮組織樣本需要立即冷凍包埋才能更好的保持樣本 RNA 質量,從而保障實驗準確性。伯豪生物自研了伯優?組織保存液,4℃條件下新鮮組織樣本離體 48 小時,細胞活性及細胞形態結果無明顯影響,有效解決樣本采集→保存運輸→冷凍包埋的不良影響。
3、高質量組織切片制備:伯豪生物科研服務團隊經大量項目樣本經驗累積,探索出 對多種類型組織制備出高質量切片方法:“不同組織類型的切片需要優化不同的條件”。
4、全面的生信分析流程和個性化數據分析。

將冷凍組織切片放置在空間轉錄組芯片的捕獲區域內,進行 HE 染色和成像后,對組織切片進行透化處理,細胞內的 mRNA 釋放,從而被芯片上帶有 oligo-dT 的探針捕獲,并且每個探針都帶有特異的位置信息(Spatial barcode),然后以 mRNA 為模版進行 cDNA 合成,構建文庫后再通過測序,獲得基因表達信息的同時,每一條測序 reads 因帶有 Spatial barcode,從而能夠獲得基因表達的位置信息。

▲圖 空間轉錄組技術的原理
注:圖片來源互聯網 - 侵刪
空間轉錄組技術包含兩種芯片,分別為組織優化芯片(Tissue Optimization)和基因表達芯片(Library Preparation)。組織優化芯片用來摸索組織透化的時間,基因表達芯片用來進行正式樣本的空間轉錄組實驗。其中基因表達芯片上有 4 個捕獲區域,每個區域大小為 6.5mm×6.5mm,每個捕獲區域中有 5000 個帶有特異地址序列的探針簇,稱為 barcoded spots,每個 spot 直徑為 55um,包含數百萬個用于捕獲的 oligo 探針序列,相鄰兩個 spot 的中心距離為 100um。探針序列的結構為:測序引物結合序列,16nt 的位置序列,12nt 的 UMI 序列以及 30nt 的 oligo-dT 序列。

▲圖 空間轉錄組兩種芯片
注:圖片來源互聯網 - 侵刪

▲圖 空間轉錄組基因表達芯片工作原理
注:圖片來源互聯網 - 侵刪

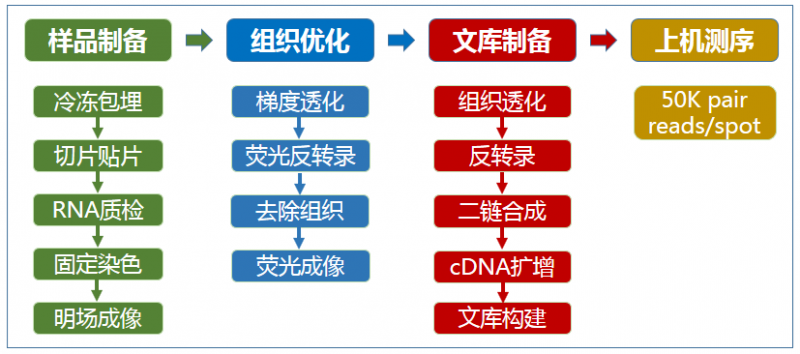
▲圖 空間轉錄組測序實驗流程

方法一:組織冷凍(不常用)
1、異戊烷和液氮浴:如圖所示,不銹鋼燒杯內倒入異戊烷至 2 / 3 體積,然后將不銹鋼燒杯放入液氮中(與異戊烷液位相同),孵育 15 分鐘。
2、新鮮樣本可用 PBS 沖洗,去除殘留血液,然后使用實驗室紙巾吸干組織表面多余的血液或液體,防止冰晶的形成(注:組織長和寬不可超過 6mm±0.2mm,否則后續實驗無法貼片)。
3、用鑷子或刮刀將組織整體浸沒在異戊烷中,直至整體冰凍(注:冷凍時間可根據組織類型和大小而改變)。
4、冷凍后,取出組織轉移到預冷的密封容器中,干冰轉移至 -80°長期保存或立即進行下一步(注:為防止組織樣品蒸發和脫水,冷凍的組織樣品必須儲存在密封容器中以長期保存)。
方法二:冷凍組織包埋(常用)
1、粉狀干冰:用研缽和杵準備干冰粉。
2、冷凍 OCT:將 OCT 放在冰上≥30 分鐘(注:OCT 請使用指定品牌)。
3、預冷鑷子:將鑷子放在干冰中≥30 分鐘。
4、包埋盒上需標記組織樣本的方向(注:在添加 OCT 和組織之前,需先在包埋盒上標記,因為一旦凍結,OCT 將迅速變成白色,這使得以后很難確定組織方向)。
5、用冷卻的 OCT 鋪平包埋盒底部,避免產生氣泡。
6、從不銹鋼燒杯中取出冰凍的組織,加入到 OCT 的包埋盒中心位置,繼續倒入 OCT 覆蓋樣本。需避免氣泡產生,尤其在組織附近。
7、立即將含有組織和 OCT 的包埋盒放在干冰粉上,直至整體凍結(約 20min 以上)。
8、將包埋盒放置到密封袋中,干冰運輸。

注意事項
1、新鮮組織樣本包埋
目前新鮮組織的包埋方法有兩種,一種是液氮 + 異戊烷法;另一種的干冰法。對于臨床手術切下來的組織樣本一般使用干冰的包埋方法。對于穿刺樣本等一些較小,較輕的樣本,一般推薦用液氮 + 異戊烷的方法進行冷凍。OCT 包埋組織塊可以在–80oC 的密封容器中長期保存,或立即進行冷凍切片。
2、冷凍切片切、組織樣本質控及貼片
由于空間轉錄組檢測的是組織中的 RNA,因此要對切片中的 RNA 質量進行檢測。我們一般取 10 片組織切片進行 RNA 抽提并質檢,確定組織中 RNA 完整性(RIN>7)。所以要求我們的組織樣本要保證可以切到至少 20 片 10um 厚度的切片,以便完成所有的實驗。
3、組織優化
組織優化的目的是摸索樣本的透化條件,保證組織切片中的 mRNA 能夠充分釋放。該步驟是獲取真實實驗結果的必要條件。否則,我們無法判斷是基因表達高低到底是因為透化不充分導致的,還是實際就是這個樣子。因此:每個樣本建議都要做透化,尤其是臨床樣本。
4、成像
應使用 Visium Imaging Test Slide 驗證成像設置。明場成像基準框和基準標記應清晰可見,并使用 Brightfeld 設置聚焦。Visium Imaging Test Slide 四個區域(A1,B2,C1,D2)具有熒光斑點,可通過 TRITC 和 Cy5 flter cubes 檢測到,熒光設置應清晰可見 A1,B2,C1 和 D2 中的熒光點,且熒光點信號應從左到右減小。
5、正式實驗
正式實驗時要對反轉錄后的 cDNA 長度分布,濃度和量進行判斷。cDNA 的長度分布在~200bp-9000bp 之間,在 1000bp 左右會有峰值(不同的組織類型會有些許差異)。
6、測序
在捕獲區域,每個組織覆蓋的 spot 建議至少測 50000 read pairs。整個捕獲區域共有 5000 個 spots。可以根據組織貼到芯片上后,覆蓋芯片的大小來判斷測序的數據量。
計算公式(Coverage Area x total spots on the Capture Area)x 50,000 read pairs/spot 例如:組織覆蓋了 60% 的區域,則數據量為(0.60 x 5,000 total spots) x 50,000 read pairs/spot=150 million read pairs。

圖 切片覆蓋芯片區域百分比

空間轉錄組數據分析的核心是根據每個芯片上每個 spot 的基因表達信息進行聚類,然后將 spot 根據坐標位置序列放回到組織的圖像上,同時可以對每個 gene 在組織上表達的空間位置進行定位。
獲得測序數據后,首先利用 Space Ranger 軟件可以自動化的對圖像進行處理、數據比對和 Barcode 處理。另外一個軟件 Loupe Cell Browser 是一個適用于 Windows 和 MacOS 的桌面應用程序,它可以快速、輕松地可視化和分析 10X Visium 數據。伯豪生物除了提供 spot 基因數和 UMI 數統計、切片 spot 聚類和聚類亞群 marker 基因分析等基礎和高級分外,同時還提供個性化分析,如特定 pathway 功能富集分析等。
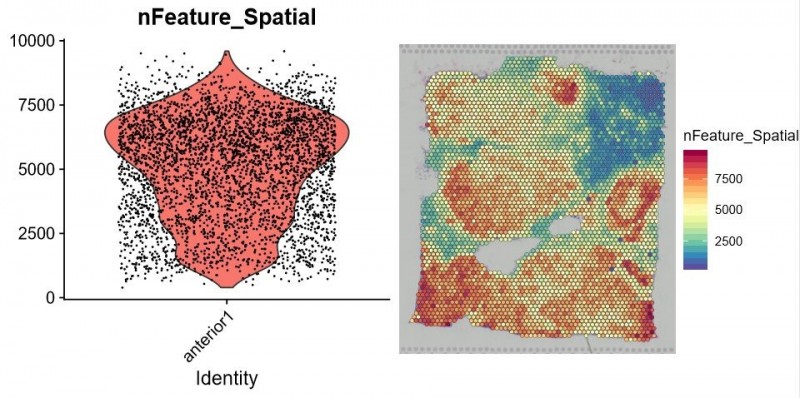
▲圖 每個 spot 特異表達的基因數統計
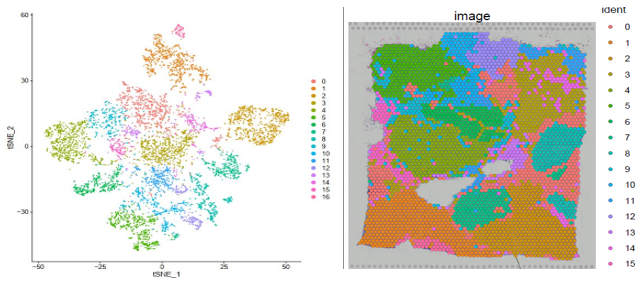
▲圖 聚類結果及切片 spot 位置分布展示

▲圖 特定 pathway 功能富集分析
結合組織區域分布對數據進行挖掘
大部分組織其實是有其特定的區域劃分的,比如說大腦里有皮層、丘腦、海馬、脈絡叢等多個區域。將組織的區域劃分和亞群(或細胞類型)的分布結合起來還是能發現很多有價值的信息的。
可以根據不同區域特異表達的 maker 基因的分布來判斷每個區域在組織切片上的位置。例如皮層 marker 基因 STX1A 的表達分布,海馬 marker 基因 HPCA 的表達分布等。
結合病理學特征對數據進行挖掘
空間轉錄組技術正真的精髓不是研究細胞亞群的分布,而在于將它在空間位置上體現的異質性跟組織病理學特征的分布進行結合,挖掘在不同病理學特征下轉錄組學的差異。這對于研究疾病病變的機制、幫助臨床實現更好的患者分子分型、以及空間位置 Biomarker 的挖掘方面都是非常有價值的。通過手動把這些區域圈出來進行轉錄組層面的比較,找出不同病灶區的特異性 marker,分析疾病在一步步發展進程中生物學功能的變化,甚至可以思考一下是否能找出一些關鍵性因子來阻斷疾病的進展。

▲圖 根據病理信息選取特定區域分析
空間轉錄組聯合單細胞 RNA 測序解析細胞類型的空間位置信息(Multimodal intersection analysis,MIA)
空間轉錄組測序可以獲得不同基因在組織切片上的空間位置信息,但不能獲得詳細的細胞類群信息(空間轉錄組不是單細胞分辨率,只能粗略的分析切片上不同位置的細胞類型)。因此,需要借助但細胞測序數據來分析細胞類型,然后通過生物信息學的分析方法將單細胞類群映射到空間轉錄組數據上。
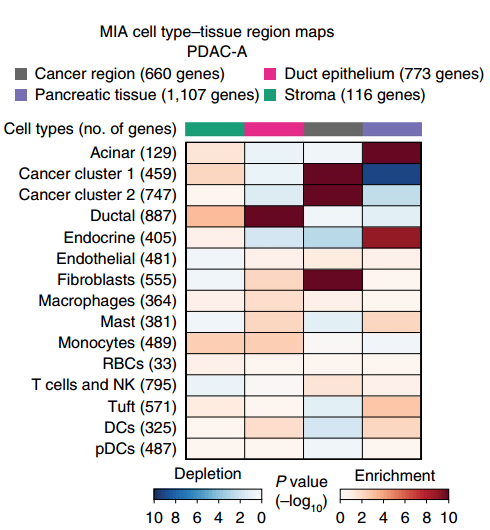
▲圖 MIA 熱圖
備注:MIA 熱圖,上方的顏色條反映了 ST 區域的子聚類(cancer region,Pancreatic tissue,Duct epithelium 和 stroma)。左側代表不同的細胞類群。色塊代表 enrichment 或者 depletion。Enrichment 代表該細胞類群富集到了該區域。Depletion 代表該細胞類群在該區域缺失。

空間位置信息,或者細胞在組織中天然的狀態在研究過程中其實具有十分重要的價值,特別針對某些研究領域,如發育生物學(不同位置的細胞接受不同的信號濃度梯度、響應不同的外界刺激,具有不同的發育命運)、腫瘤生物學(腫瘤組織與癌旁組織的區別,腫瘤細胞侵潤過程中腫瘤細胞的變化與對正常細胞的影響,腫瘤轉移的不同過程階段等)、腦神經科學(不同腦區位置的神經元結構、神經連結,中間神經元投射,突觸前后,神經膠質相互影響等等),細胞來源的位置信息是極為關鍵的決定因素。
空間轉錄組測序技術除了在人和動物上得到了廣泛的應用之外,在植物領域也有所突破。2017 年發表在 nature plant 上的一篇文章就闡釋了空間轉錄組在擬南芥中的應用,利用空間數據作者分析了 141 個基因的表達水平差異,8 個花序組織域中 189 條通路。伯豪生物作為較早的空間轉錄組測序技術服務商,也在植物的空間轉庫組領域有所突破。開發了多種植物組織的空間轉錄組樣本制備,透化條件摸索等。獲得了寶貴的項目經驗。
常見空間轉錄組的應用方向主要在腫瘤學,免疫學,發育生物學,神經科學及病理學等方向。
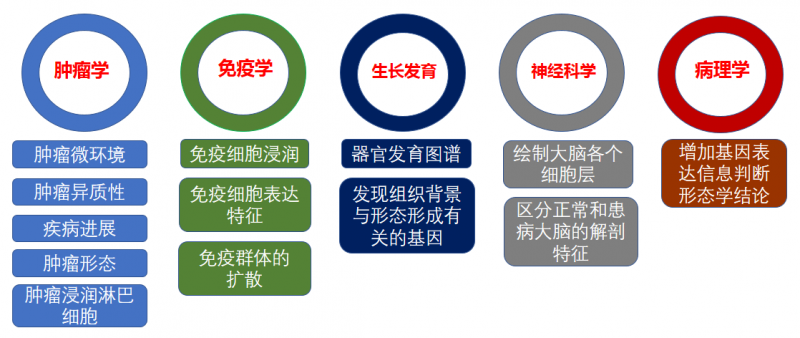
▲圖 空間轉錄組的應用方向
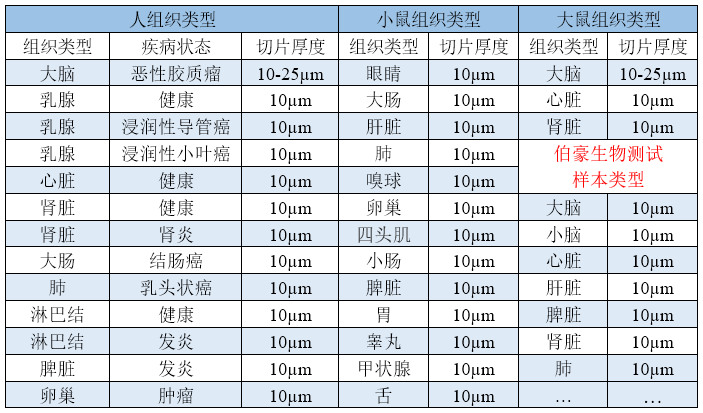
▲表 已做過優化的樣本類型

案例一
空間轉錄組學聯合單細胞 RNA-seq 揭示胰腺導管腺癌的組織結構
Integrating microarray-based spatial transcriptomics and single-cell RNA-seq
reveals tissue architecture in pancreatic ductal adenocarcinomas [5]
發表雜志:Nature biotechnology
影響因子:36.558
發表時間:2020 年 1 月
單細胞 RNA 測序(single RNA sequence,scRNA-seq) 能夠系統地識別組織中的細胞類群,但它不能獲取各個細胞類型在組織中的空間位置信息。空間轉錄組測序(spatial transcriptomics,ST),可以獲得不同基因在組織切片上的空間位置信息,但不能獲得詳細的細胞類群信息。作者通過對胰腺導管癌病人的同一樣本同時進行 scRNA 測序和 ST 測序。同時開發了一種多模態交叉分析方法(Multimodal intersection analysis,MIA)來將單細胞測序數據映射到空間轉錄組數據上,獲得各種細胞類型在組織上的空間分布。研究發現導管細胞、巨噬細胞、樹突狀細胞和癌細胞的亞群在不同的空間位置區域上富集或丟失,以及與其他類型的細胞有顯著的共富集。此外,作者還發現了炎癥成纖維細胞和表達應激反應基因模塊的癌細胞的定位。
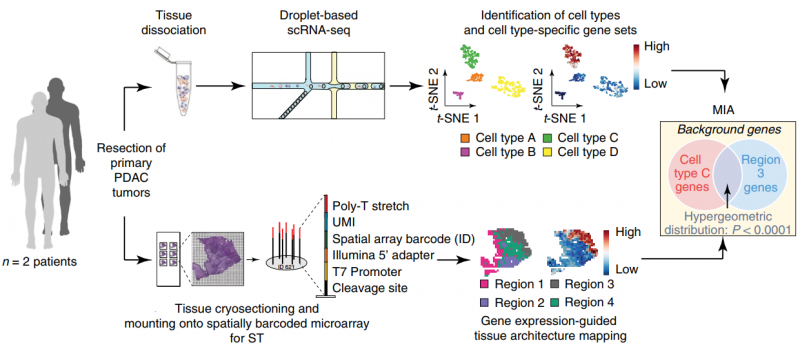
圖:ST 聯合 scRNA 揭示胰腺導管癌組織細胞類型及結構
案例二
空間轉錄組聯合單細胞測序揭示心臟發育
A Spatiotemporal Organ-Wide Gene Expression and Cell Atlas of the Developing Human Heart[11]
發表雜志:Cell
影響因子:38.637
發表時間:2019 年 12 月
摘要:人類心臟形態發生的過程尚不清楚。它的完整特性需要用單細胞空間分辨率深入探索器官范圍內基因表達的協調。在這里,我們提出了一種分子方法,揭示了在三個發育階段的胚胎心臟細胞類型的轉錄景觀,并將細胞類型特異性基因表達映射到特定的解剖結構域。空間轉錄組學鑒定了在每個發育階段對應不同解剖區域的獨特基因譜。作者通過對人類心臟發育有三個階段:孕早期 4.5- 5 周,6.5 周和懷孕后 9 周的人類胚胎心臟樣品進行單細胞 RNA 測序鑒定的人類胚胎心臟細胞類型,同時通過空間轉錄組測序技術來獲取基因表達的空間位置信息。然后使用原位測序來細化這些結果,并為三個發育階段創建一個空間亞細胞圖譜。形成了一個公開的人類心臟發育的網絡資源,以促進未來對人類心臟發生的研究。
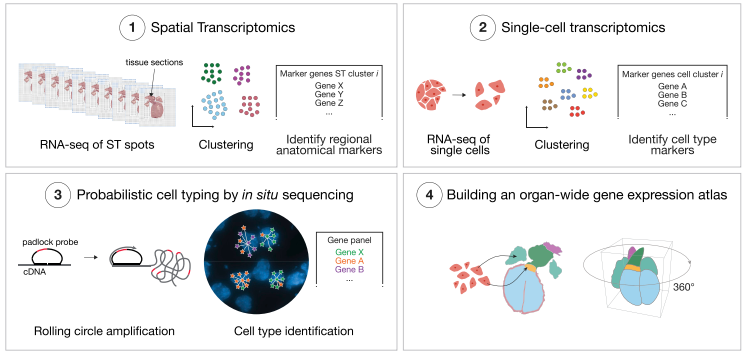
圖 18 實驗設計思路
A、本研究包括三種人類心臟組織的發育階段。B、本研究所采用的分子生物學方法:(1)利用空間轉錄組技術(ST),鑒定出了解剖學區域特異的 marker;(2)用 scRNA-seq 技術分析心臟中間時間點的細胞類型異質性;(3)用 ISS 技術定位亞細胞分辨率的關鍵基因;(4)構建器官水平的 3D 基因表達圖譜。
案例三
單細胞 RNA 測序 & 空間轉錄組測序“強強聯手”揭示腸道發育
Spatiotemporal analysis of human intestinal development at single-cell resolution
發表期刊:Cell
影響因子:38.637
發表時間:2021 年 1 月
腸道是人體大的屏障器官,與腸道微生物共生協調營養需求和免疫。多種相互關聯的細胞類型構成了成熟的腸道及其不同的形態,但其發育的分子基礎仍不清楚。來自牛津大學 Simmons 和 Koohy 教授團隊通過單細胞測序聯合空間轉錄組技術來研究腸道發育過程中形態的變化。研究對來自 17 例胚胎的 77 個樣本進行了單細胞 RNA 測序(使用寡核苷酸標記抗體的多重混樣技術)以及對來自 5 個樣本的 8 張切片進行了空間轉錄組測序。通過一系列的生物信息學分析方法,包括細胞間通訊網絡構建,SCENIC 轉錄因子模塊分析,擬時序分析,RNA 速率分析,基因功能富集分析等,共鑒定了 101 種細胞類型,包括上皮細胞和間充質祖細胞群和與關鍵形態發生的重要程序。作者描述了隱窩 - 絨毛軸形成的原理,發育中的腸道的神經、血管、間充質形態發生和免疫群體。確定了發育中的成纖維細胞和肌成纖維細胞亞型的分化層次,并描述了它們的不同功能,包括作為血管生態位細胞的功能。作者確定了 Peyer’s patches 和 gut-associated lymphoid tissue (GALT) 的起源,并描述了位置特異性免疫程序。提出了一個無偏倚的分析形態梯度,可以用來指引連續的細胞分化、定義細胞、以及罕見的發育性腸道疾病相關的位置區域。此外,作者還編制了一個公開的胎兒腸道在時間和空間層面發育(STAR-FINDer) 的在線數據庫,以促進進一步的工作。
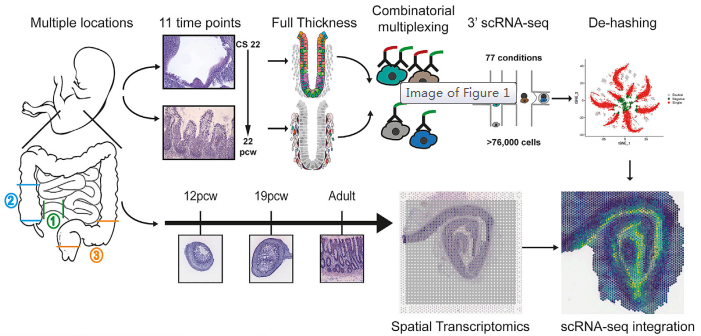
圖 研究概況
案例四
空間轉錄組測序揭示阿爾茲海默疾病機制
Spatial Transcriptomics and In Situ Sequencing to Study Alzheimer’s Disease
發表期刊:Cell
影響因子:38.637
發表時間:2020 年 8 月
雖然在阿爾茨海默病(AD) 淀粉樣斑塊周圍觀察到復雜的炎癥樣改變,但對這種反應的分子變化和細胞相互作用知之甚少。在 AD 小鼠模型中,作者利用空間轉錄組學技術研究淀粉樣斑塊周圍直徑為 100 毫米的組織結構域發生的轉錄變化。研究證實了髓磷脂和少突膠質細胞基因(OLIGs) 富集的基因共表達網絡的早期改變,而涉及補體系統、氧化應激、溶酶體和炎癥的斑塊誘導基因(豬)的多細胞基因共表達網絡在疾病的后期顯著。此外,在小鼠和人類大腦切片上使用原位測序確認了大多數在細胞水平上觀察到的改變。空間轉錄組學分析為解開 AD 和其他腦部疾病致病特征附近的失調細胞網絡提供了一種新的方法。
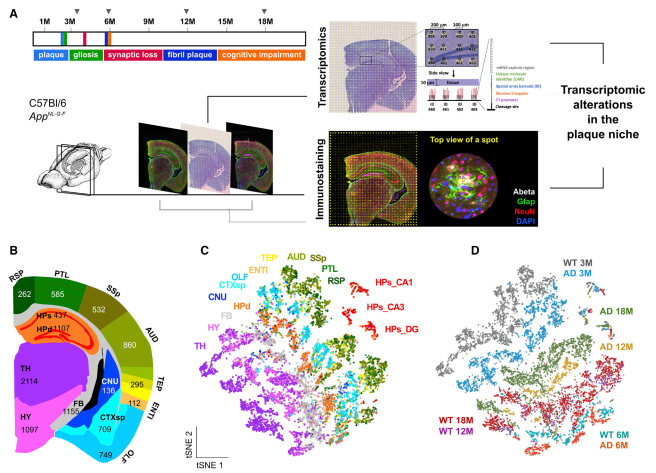
圖 成年 AD 小鼠大腦的空間解析轉錄組譜
案例五
空間轉錄組圖譜為前列腺癌提供異質性的新視圖
Spatial Maps of Prostate Cancer Transcriptomes Reveal an Unexplored
Landscape of Heterogeneity
發表期刊:Nature communication
影響因子:12.212
發表時間:2018 年 9 月
前列腺癌包含大量的腫瘤內異質性,在原發腫瘤和遠距離轉移中都存在基因改變前列腺癌的病理嚴重程度,盡管有分子標記和核磁共振的進展,但通常是根據 Gleason 分級(Gs)系統來評分的,該系統僅使用組織學數據,經常在血液和腫瘤分期中補充 PSA 測量。然而,這種分類方法有局限性,并提出了新的備選方案。利用一種新的反卷積方法,作者分析了近 6750 個組織區域的轉錄組,并提取了不同組織成分的不同表達譜,如間質、正常和針腺、免疫細胞和癌癥。同時區分了健康區域和病變區域,從而對前列腺癌進展過程中的基因表達變化提供了見解。與病理學家的注釋相比,空間轉錄組可以更準確地描述了癌灶的范圍,有趣的是,與組織學變化無關。
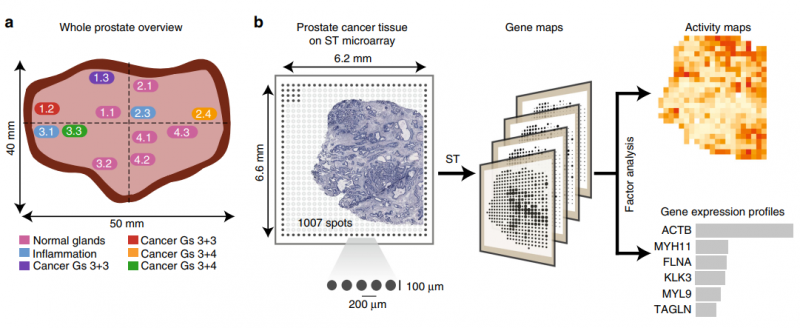
圖 前列腺癌的空間轉錄組學(ST) 研究設計
案例六
空間轉錄組解析模式植物組織基因表達的空間信息
Spatially resolved transcriptome profiling in model plant species
發表期刊:Nature plant
影響因子:13.256
發表時間:2017 年 5 月
要理解復雜的生物系統,需要對特定組織域進行功能研究。然而,現有的空間轉錄組測序技術僅適用于有限范圍的生物,主要是哺乳動物。在這里,作者提出了可用與植物組織的方法,在空間分辨率的狀態下來廣泛的解析模式植物系統。該過程包括高通量的空間轉錄組分析,然后是空間基因和通路分析。作者首先從模型被子植物和裸子植物的顯微切片中生成空間轉錄組譜,證明了該技術的可行性。在擬南芥中,利用空間數據分析了 8 個花序組織結構域中 141 個基因和 189 條通路的表達水平差異。作者通過將空間轉錄組學技術和功能譜分析技術相結合這一新策略,應用于廣泛的植物物種,該技術將是一種解決發育和進化生物學基本問題的關鍵方法。
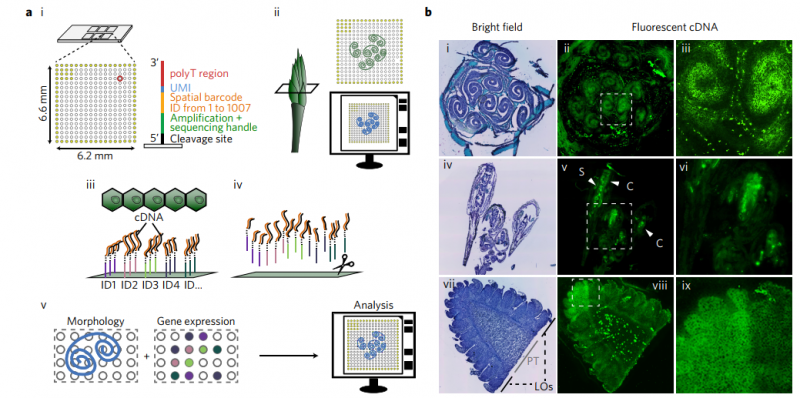
▲圖 植物的空間解析轉錄組分析

[1]. St?hl PL, Salmén F, Vickovic S, et al. Visualization and analysis of gene expression in tissue sections by spatial transcriptomics. Science 2016, 353(6294):78-82.
[2]. Peng G, Suo S, Chen J, et al. Spatial Transcriptome for the Molecular Annotation of Lineage Fates and Cell Identity in Mid-gastrula Mouse Embryo. Dev Cell 2016, 36(6):681-697.
[3]. https://www.spatialomics.org/SpatialDB/
[4]. Michaela Asp, Stefania Giacomello, et al. A Spatiotemporal Organ-Wide Gene Expression and Cell Atlas of the Developing Human Heart[J]. Cell, 2019, 179, 1647–1660.
[5]. Maniatis S, ?ij? T, Vickovic S, et al. Spatiotemporal dynamics of molecular pathology in
amyotrophic lateral sclerosis[J]. Science, 2019, 364(6435): 89-93.
[6]. Carlberg K, Korotkova M, Larsson L, Catrina AI, Stahl PL, Malmstrom V. Exploring inflammatory signatures in arthritic joint biopsies with Spatial Transcriptomics[J]. Scientific reports. 2019,9(1):18975.
[7]. José, Fernández, Navarro, et al. ST viewer: a tool for analysis and visualization of spatial transcriptomics datasets.[J]. Bioinformatics (Oxford, England), 2019.
[8]. Berglund E , Maaskola J , Schultz N , et al. Spatial maps of prostate cancer transcriptomes reveal an unexplored landscape of heterogeneity[J]. Nature Communications, 2018, 9(1).
[9]. ST Spot Detector: a web-based application for automatic spot and tissue detection for spatial Transcriptomics image datasets[J]. Bioinformatics, 2018.
[10]. Anna L , Natalija G , Tove B , et al. Gene expression profiling of periodontitis-affected gingival tissue by spatial transcriptomics[J]. Scientific Reports, 2018, 8(1):9370.
[11]. Kim T , Hanna E , Jonas M , et al. Spatially resolved transcriptomics enables dissection of genetic heterogeneity in stage III cutaneous malignant melanoma[J]. Cancer Research, 2018:canres.0747.2018.
[12]. Salmen F, Stahl PL, Mollbrink A, Navarro JF, Vickovic S, Frisen J, et al. Barcoded solid-phase RNA capture for Spatial Transcriptomics profiling in mammalian tissue sections[J]. Nature protocols. 2018,13(11):2501-34.
[13]. Giacomello S , Fredrik Salmén, Terebieniec B K , et al. Spatially resolved transcriptome profiling in model plant species[J]. Nature Plants, 2017, 3(6):17061.
[14]. Asp M , Salmén, Fredrik, St?Hl P L , et al. Spatial detection of fetal marker genes expressed at low level in adult human heart tissue[J]. Scientific Reports, 2017, 7(1):12941.
[15]. Stahl P L , Salmen F , Vickovic S , et al. Visualization and analysis of gene expression in tissue sections by spatial transcriptomics[J]. Science, 2016, 353(6294):78-82.
[16]. Vickovic S , St?Hl P L , Salmén, Fredrik, et al. Massive and parallel expression profiling using microarrayed single-cell sequencing[J]. Nature Communications, 2016, 7:13182.
[17]. Anders, Jemt, Fredrik, et al. An automated approach to prepare tissue-derived spatially barcoded RNA-sequencing libraries.[J]. Scientific reports, 2016.
[18] Berglund E , Maaskola J , Schultz N , et al. Spatial maps of prostate cancer transcriptomes reveal an unexplored landscape of heterogeneity[J]. Nature Communications, 2018, 9(1).
[19] Chen W T , Lu A , Craessaerts K , et al. Spatial Transcriptomics and In Situ Sequencing to Study Alzheimer's Disease[J]. Cell, 2020.
[20] Giacomell S , Fredrik Salmén, Terebieniec B K , et al. Spatially resolved transcriptome profiling in model plant species[J]. Nature Plants, 2017.
[21] David Fawkner-Corbett, et al. Spatiotemporal analysis of human intestinal development at single-cell resolution[J]. Cell. 2021 Feb 4;184(3):810-826.e23. doi: 10.1016/j.cell.2020.12.016. Epub 2021 Jan 5.

| 序號 | 文件類型 | 查閱 |
| 1 | 【畫冊】空間基因表達解決方案 | 點擊查看 |
| 2 | 【畫冊】石蠟樣本(FFPE)空間基因表達解決方案 | 點擊查看 |
| 3 | 【畫冊】新鮮樣本(FF)空間基因表達解決方案 | 點擊查看 |


