 |  |

人類基因組測序開展的數十年以來,研究人員建立了遺傳因素與許多 Jí 病之間的聯系。但是,編碼 基因組 DNA 的核苷酸只是遺傳因素中影響細胞功能和整體健康的一部分,表觀遺傳學(指在某些情況下 不修改 DNA 序列而發生的可遺傳的改變)也具有重要功能。DNA 甲基化是一種已經過大量研究的表觀遺傳學標記,其可化學修飾胞嘧啶和腺嘌呤。胞嘧啶甲基化常發生在基因組中的 CG 序列,稱為 CpG 位點,它以細胞特異性的方式廣泛地調控基因表達。全基因組關聯研究 (GWAS) 將 DNA 甲基化的改變與復雜病例如癌癥和肥胖以及復雜的生物學狀態例如衰老和發育聯系起來 [1,2]。近年來甲基化測序已擴展到基因表達之外,開始在 Jí 病診斷中獲得關注 [1,3-5]。近期研究表明差異甲基化是癌癥檢 Cè 中信息豐富且 靈敏的標記,不論甲基化是否與基因表達相關 [6]。
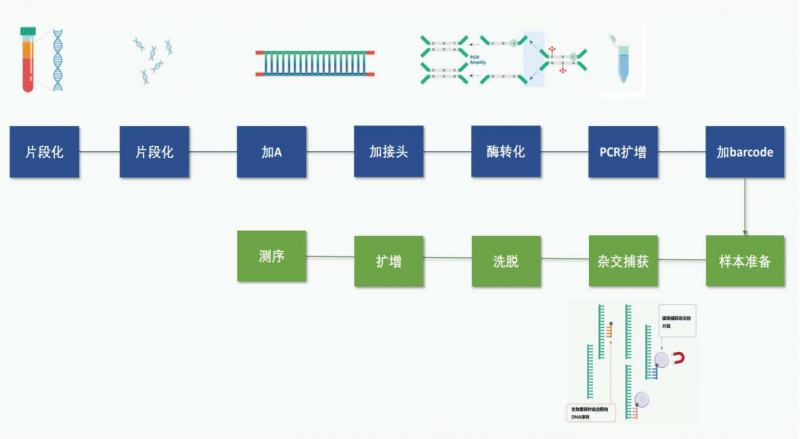
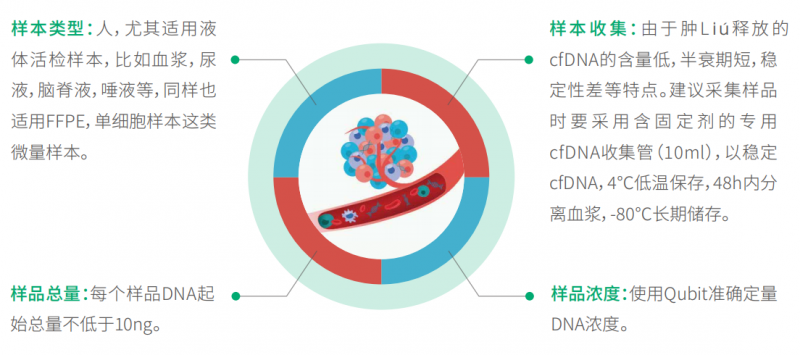
目前,DNA 甲基化在癌癥液體活檢(如 cfDNA)中已經有很多相關研究,并受到越來越多的關注。常 見的 DNA 甲基化研究手段都是基于亞硫酸氫鹽(化學試劑)的轉化,這種方法很劇烈,對 DNA 損傷嚴重,對 DNA 起始量有著較高要求。而血液 cfDNA 的含量通常很低,只有幾十 ng,普通的亞硫酸氫鹽測序手段很難滿足。因此伯豪生物引進 EM-seq(Enzymatic methyl sequencing),該技術基于酶學溫和轉化,可實 現 DNA 甲基化位點的單堿基分辨率,適用于癌癥早篩液體活檢樣本的標志物研究(如:血漿 cfDNA,尿液 cfDNA,腦脊液 cfDNA 等)也適用于研究細胞分化的單個細胞超微量樣本(如:卵母細胞,精母細胞,胚胎等)。EM-seq 提供了穩健的全流程完整解決方案,用于識別人類基因組中的甲基化區域。文庫制備過程 中,采用獨特的酶促轉化,它對脫氧核糖核酸(DNA) 的損害小得多,需要的樣本輸入更少,從而獲得更高 質量、性能更好的文庫。Twist 自定義甲基化探針組合設計為 CpG 檢 Cè 提供了有效特別的探針,用于靶向富集。優化的雜交試劑增加了工作流程的時間靈活性,并提高了中靶率。甲基化測序涉及酶或化學方法,這些方法可使未甲基化的胞嘧啶通過一系列反應,通過脫氨基 轉化為尿嘧啶,而甲基化的胞嘧啶則 保持完整(圖 1)。在擴增過程中,尿嘧啶對應的互補鏈上會互補配對加上腺嘌呤,造成未甲基化的胞嘧啶的原始位置引入胸腺嘧啶。如圖所示序列終產物是不對稱的,轉化后形 成兩條不同的雙鏈 DNA 分子(圖上);對于甲基化的 DNA,同樣的過程則產生另外不同序列(圖下)。
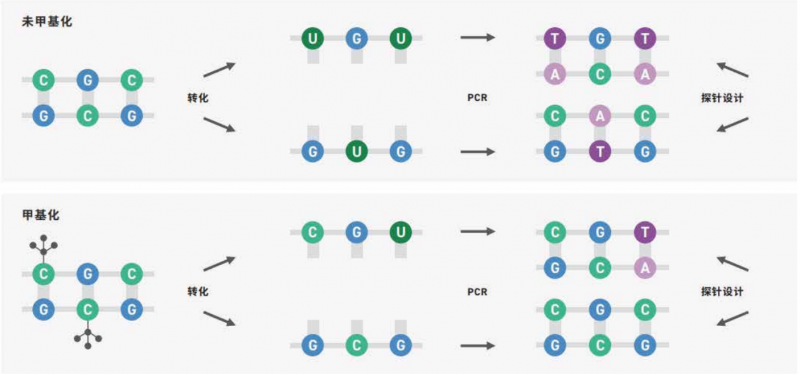
▲圖 1

產品優勢
1、酶轉化 Zuì大限度降低 DNA 損壞,超低 DNA 起始量要求,低至 10ng;
2、卓越的比對率,GC 均一性;
3、高靈敏度,分辨率可達到單個堿基;
4、相比亞硫酸氫鹽方法檢 Cè 可多檢 Cè 15% 的甲基化位點;
5、靈活適用不同靶向區域。
目錄化產品
人(hg38)的 134Mb 設計長度(覆蓋約 400 萬 CpG 位點),基于新的 UCSC, Ensembl, ENCODE 等新的數據庫,關注那些已知甲基化可影響基因調節的區域:57% CpG open seas (interCGI), 21% CpG island shores, 15% CpG islands, 8% CpG island shelves。


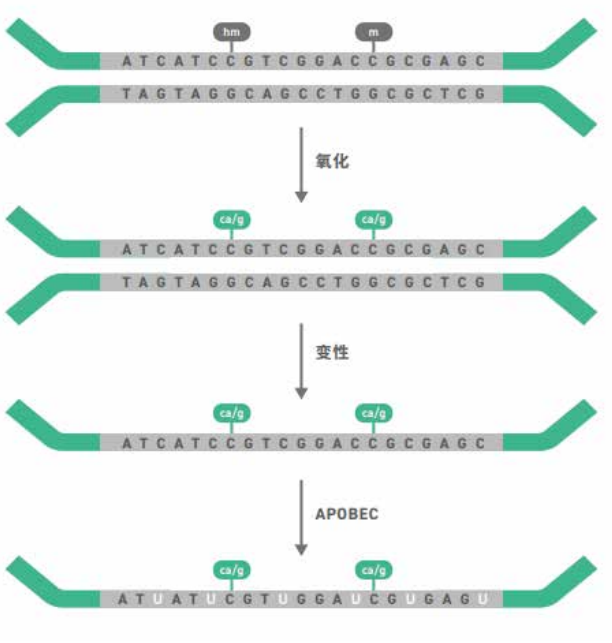
▲圖。EM-seq 轉化涉及一系列酶反應,以識別未甲基化胞嘧啶。
在 Dì 一次反應期 間,10-11 易位雙加氧酶 2(TET2) 將甲基 化胞嘧啶(5mC 和 5hmC)轉化為 5 - 羧基 胞嘧啶(5caC) 以及氧化增強劑葡糖苷 酸 5hmC(5ghmC)。這些反應保護 5mC 和 5hmC 免受下游脫氨作用。然后,在 APOBEC 將胞嘧啶脫氨至尿嘧啶之前, 使 DNA 變性。隨后的聚合酶鏈式反應 (PCR) 擴增將修飾的 5mC 或 5hmC 轉化 為胞嘧啶,并將尿嘧啶轉化為胸腺嘧啶。PCR 后,核酸序列與重亞硫酸鹽轉化的 序列一致,使得 EM-seq 與現有分析流程 (如 Bismark 和 bwa-meth)相兼容。
酶法的高效轉化
| 指標 | 末甲基化 Lambda DNA | CpG 甲基化 pUC19 DNA |
| 預期轉化效率 | ≥99.5% | ≥99.5% |
| 實測轉化效率 | 99.77% | 99.57% |
| 預期 CpG 甲基化水平 | 約 0.5% | 95-98% |
| 實測 CpG 甲基化水平 | 0.22228% | 95.7572% |
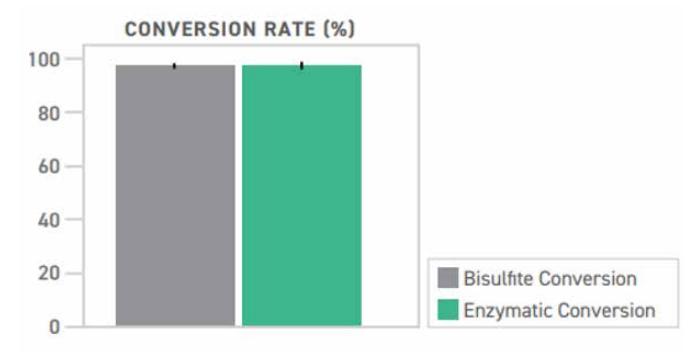
在 EM-seq 和 BS-seq 中,酶和亞硫酸氫鹽都是將未甲基化的胞嘧啶轉化為胸腺嘧啶,且轉化效率接近。選擇 CpG 甲基化的 pUC19 DNA 和未甲基化的 Lambda DNA 這種已知甲基化 DNA 作為質控對象,結果顯示,兩種文庫轉換方法的轉化率均達到 99.5%,以胞嘧啶在非 CpG 位點轉化為胸腺嘧啶的百分比來測量。





1、Jones PA. Functions of DNA methylation: islands, start sites, gene bodies and beyond. Nat Rev Genet 2012; 13: 484-492.
2、Yang X, Shao X, Gao L, Zhang S. Comparative DNA methylation analysis to decipher common and cell type-specifific patterns among multiple cell types. Brief Funct Genomics 2016; 15: 399-407.
3、Loh M, Zhou L, Ng HK, Chambers JC. Epigenetic disturbances in obesity and diabetes: Epidemiological and functional insights. Mol Metab 2019; 27S: S33-S41.
4、Ferrucci L, Gonzalez-Freire M, Fabbri E et al. Measuring biological aging in humans: A quest. Aging Cell 2020; 19: e13080.
5、Perez RF, Santamarina P, Tejedor JR et al. Longitudinal genome-wide DNA methylation analysis uncovers persistent early-life DNA methylation changes. J Transl Med 2019; 17: 15.
6、Locke WJ, Guanzon D, Ma C et al. DNA Methylation Cancer Biomarkers: Translation to the Clinic. Front Genet 2019; 10: 1150.
7、Liu MC, Oxnard GR, Klein EA et al. Sensitive and specifific multi-cancer detection and localization using methylation signatures in cell-free DNA. Ann Oncol2020; 21: 745-759.
8、Vaisvila R, Ponnaluri VKC, Sun Z et al. Enzymatic methyl sequencing detects DNA methylation at single-base resolution from picograms of DNA. Genome Res 2021.

