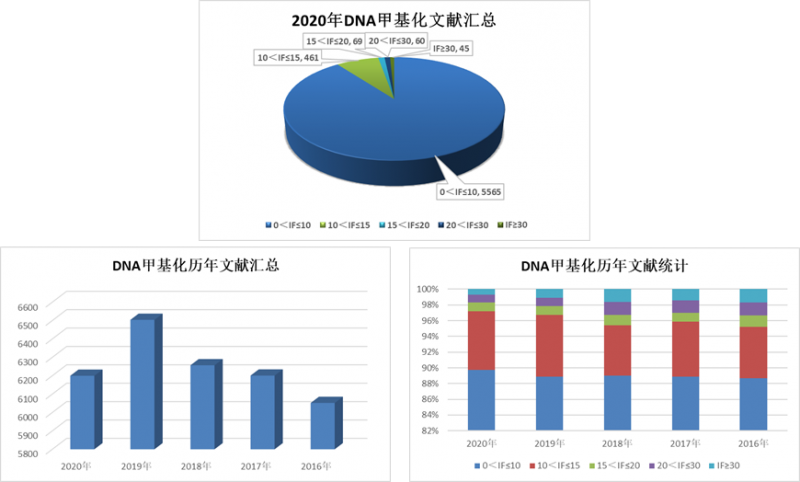
在剛剛過去的 2020 年中,以 DNA methylation 為關(guān)鍵詞在 Pubmed 中共能檢索到 6200 篇文章。其中,IF>10 的文章共計 635 篇,IF>20 的共計 105 篇,IF>30 的共計 45 篇。和前五年呈現(xiàn)出的顯著遞增趨勢相比,2020 年所發(fā)表文獻的數(shù)量有明顯下降,高分文獻的占比有所減少,這和去年的新冠疫情影響應(yīng)該有很大關(guān)系。
雖然如此,2020 年 DNA 甲基化研究的高分文獻也有很多值得研讀的,小編也在這里精挑細選了幾篇不同研究思路的高分甲基化文章(IF>20)和大家重點分享。
一、DNA 甲基化多中心研究(中介效應(yīng)研究)
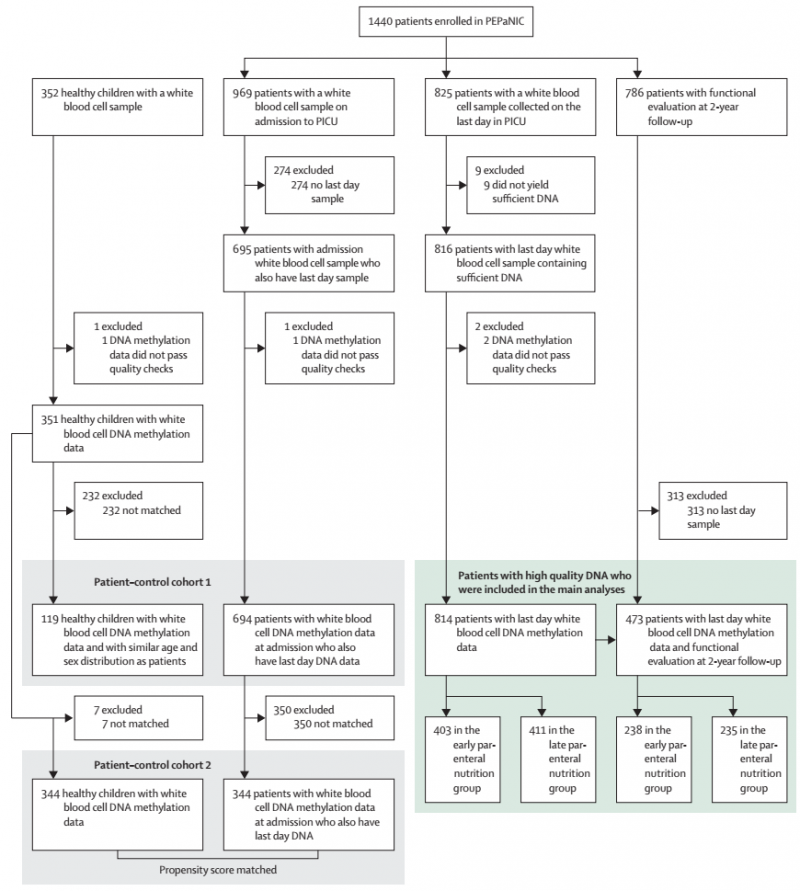
2020 年 2 月 18 日,比利時天主教魯汶大學(xué)醫(yī)院、荷蘭鹿特丹伊拉斯謨大學(xué)醫(yī)學(xué)中心索菲亞兒童醫(yī)院和加拿大阿爾伯塔大學(xué)斯托雷兒童醫(yī)院在《Lancet Respir Med》(IF 25.094)聯(lián)合發(fā)表“Effect of early parenteral nutrition during paediatric critical illness on DNA methylation as a potential mediator of impaired neurocognitive development: a pre-planned secondary analysis of the PEPaNIC international randomised controlled trial”——“PIUC(兒童重癥監(jiān)護病房)患者早期 PN(早期補充腸外營養(yǎng))以 DNA 甲基化為潛在介質(zhì)對神經(jīng)認知發(fā)育受損的影響”。

作者在 17 年同篇發(fā)表的文章中,通過大型隨機對照實驗 PEPaNIC 發(fā)現(xiàn)早期 PN 會對兒童神經(jīng)認知發(fā)育產(chǎn)生負面影響。這項針對多中心 PEPaNIC 試驗(2012-2018)的分析主要包括收集到在 PICU 后一天患者的血液樣本(n = 825,PICU 入院時年齡為 0 -17 歲),其中早期 PN 和晚期 PN 的患者各占一半,并將這些樣本與相匹配的 352 位健康兒童進行比較。結(jié)果發(fā)現(xiàn)和健康兒童相比,PICU 住院患者存在 159 個差異 CpG 位點甲基化位點,這些差異甲基化的 CpG 位點主要位于大腦發(fā)育,可塑性和信號傳導(dǎo)等相關(guān)的基因上。同時,作者使用多元線性和非線性回歸分析來評估早期 PN 與晚期 PN 對疾病引起的 DNA 甲基化改變的影響,以及 CpG 位點甲基化水平可以在多大程度上解釋 2 年后神經(jīng)認知發(fā)育受損的程度。結(jié)果發(fā)現(xiàn),早期 PN 中的氨基酸劑量會導(dǎo)致其中 37 個差異 CpG 位點的甲基化差異,說明早期 PN 中的氨基酸劑量是對影響神經(jīng)認知發(fā)育的主要因素。
參考文獻 DOI: 10.1016/S2213-2600(20)30046-1
二、癌癥 ctDNA 甲基化標志物研究
循環(huán)腫瘤 DNA (Circulating tumor DNA, ctDNA) 是腫瘤細胞破裂掉落下來的 DNA 片段,進入了外周血循環(huán)系統(tǒng)。ctDNA 攜帶了腫瘤基因組一定的突變信息,還攜帶甲基化 5mC/ 羥甲基化 5hmC 等信息,在腫瘤早篩早診等方面具有獨特的應(yīng)用價值,是液體活檢中的重要分支。
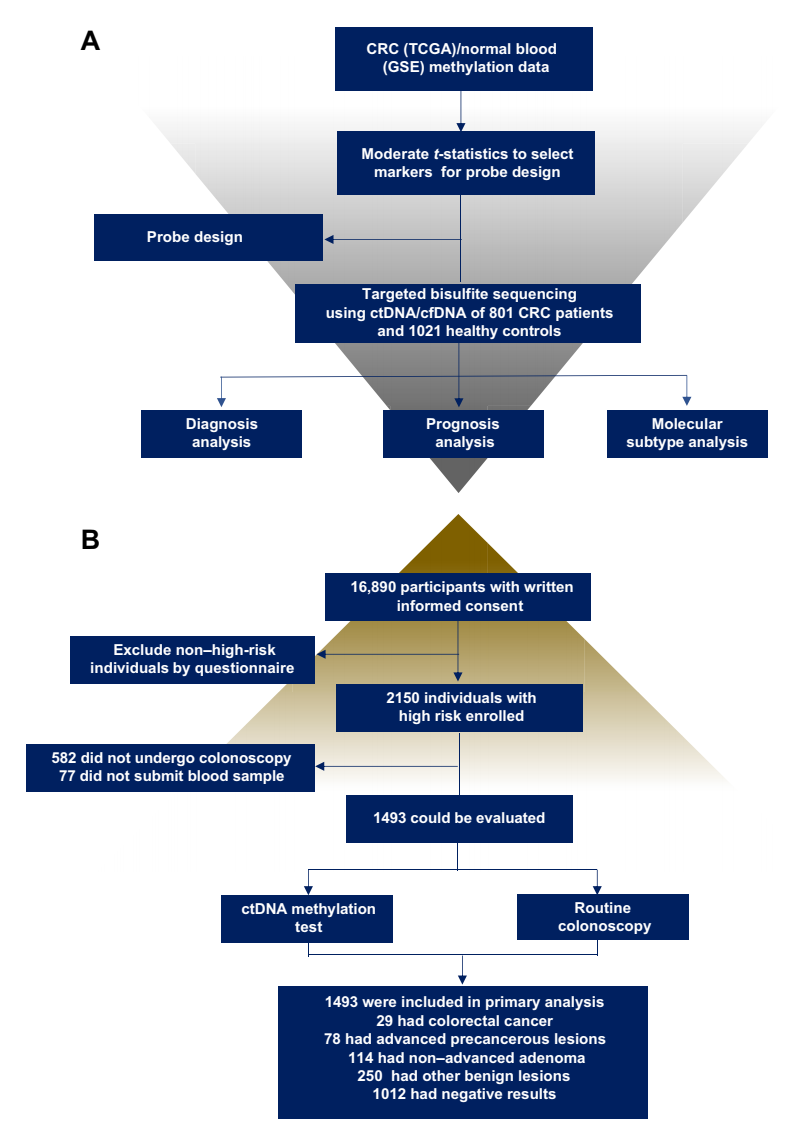
中山大學(xué)腫瘤防治中心徐瑞華教授課題組在《Sci Transl Med》(IF 16.304)上發(fā)表“Circulating tumor DNA methylation profiles enable early diagnosis, prognosis prediction, and screening for colorectal cancer”——“應(yīng)用 ctDNA 甲基化標志物可進行結(jié)直腸癌(CRC)的早期診斷,預(yù)后預(yù)測和篩查”,這是繼 17 年發(fā)表在《Nature materials》(IF 38.663)肝癌 ctDNA 甲基化標志物研究后的又一篇佳作。

作者首先基于公共數(shù)據(jù)庫篩選到 544 個 CpG 位點,并利用探針捕獲測序的方法在 801 例 CRC 患者 /1021 例健康人群血漿中提取的 cfDNA 進行了訓(xùn)練,篩選到 9 個 CpG 位點構(gòu)建診斷模型,訓(xùn)練集和驗證集中該模型診斷率高度一致(AUC=0.96),且準確性明顯高于目前臨床上使用的腸癌血檢標志物——癌胚抗原(CEA)(AUC=0.67)。同時,作者篩選到 5 個 CpG 位點用于構(gòu)建預(yù)后模型,將得到的預(yù)后評分指數(shù)(cp-score)聯(lián)合目前常用的臨床預(yù)后指標,如腫瘤原發(fā)部位、TMN 分期、CEA 等,能明顯增強預(yù)后預(yù)測的準確性。接著,作者分別分析了診斷模型中的 9 個 CpG 位點,其中 cg10673833 位點的 AUC 為 0.91,在前瞻性隊列(n=1493)中驗證該位點的對腫瘤的檢出敏感性達 89.7%,特異性達 86.8%,對進展期腺瘤的檢出率敏感性達 33.3%,敏感性和特異性均較現(xiàn)有的無創(chuàng)篩查方法有所提高。
參考文獻 DOI: 10.1126/scitranslmed.aax7533
2020 年 06 月 12 日,《Nature Medicine》(IF 36.13)同期發(fā)表了兩篇文章使用 cfMeDIP-seq 技術(shù)分別尋找腎細胞癌和顱內(nèi)腫瘤 ctDNA 甲基化標志物的文章,小編就不再這里再進行一一介紹了,感興趣的小伙伴可以通過下邊的 DIO 號獲取或者聯(lián)系我們直接獲得全文哦~
參考文獻:
Nuzzo P V , Berchuck J E , Korthauer K , et al. Detection of renal cell carcinoma using plasma and urine cell-free DNA methylomes[J]. Nature medicine, 2020, 26(7).
Nassiri F , Chakravarthy A , Feng S , et al. Detection and discrimination of intracranial tumors using plasma cell-free DNA methylomes[J]. Nature medicine, 2020, 26(7).
三、DNA 甲基化多組學(xué)研究
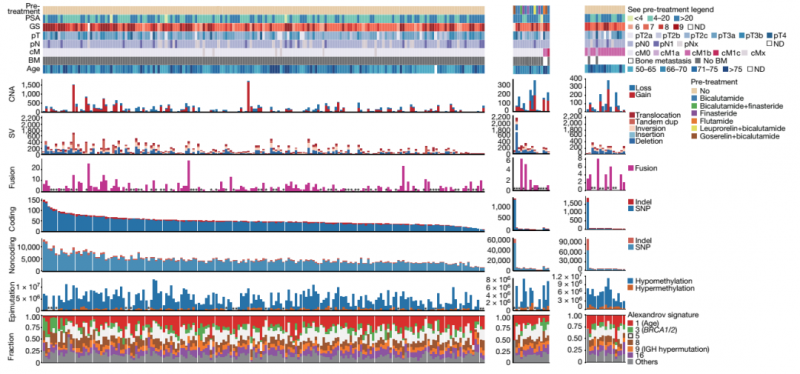
2020 年 3 月 25 日,海軍軍醫(yī)大學(xué)長海醫(yī)院孫穎浩院士領(lǐng)銜在《Nature》(IF 42.778)發(fā)表“A genomic and epigenomic atlas of prostate cancer in Asian populations”,揭示了中國前列腺癌人群特有的多組學(xué)分子特征。

作者通過對 208 例原發(fā)性前列腺癌的癌組織與癌旁組織進行 WGS、WGBS、RNA-seq 和 miRNA-seq 檢測,分別從 CAN、SV、融合基因、編碼區(qū)和非編碼區(qū)的 SNV/Indel、胚系表觀突變多個水平展現(xiàn)中國人群前列腺癌基因組和表觀基因組圖譜(CPGEA)。同時,在此基礎(chǔ)上,作者將該數(shù)據(jù)與 2554 例西方患者比較發(fā)現(xiàn),中國患者的基因組突變與西方患者存在明顯差別,這為中國前列腺癌人群的個性化治療提供了理論依據(jù)。
參考文獻 DOI: 10.1038/s41586-020-2135-x
2020 年 7 月 13 日,密歇根大學(xué)在《Nat Genet》(IF 27.603)發(fā)表了關(guān)于前列腺轉(zhuǎn)移癌的全基因組與表觀基因組圖譜的研究成果——“The DNA methylation landscape of advanced prostate cancer”,該項目使用了西方人群的大隊列進行研究,感興趣的小伙伴可以聯(lián)系我們即可輕松獲取文獻哦~
參考文獻:
Gagliardi A , Porter V L , Zong Z , et al. Analysis of Ugandan cervical carcinomas identifies human papillomavirus clade–specific epigenome and transcriptome landscapes[J]. Nature Genetics.
Zhao S G , Chen W S , Li H , et al. The DNA methylation landscape of advanced prostate cancer[J]. Nature Genetics, 2020, 52(8).
Carrillo-Reixach J , Torrens L , Simon-Coma M , et al. Epigenetic footprint enables molecular risk stratification of hepatoblastoma with clinical implications - ScienceDirect[J]. Journal of Hepatology, 2020, 73(2):328-341.
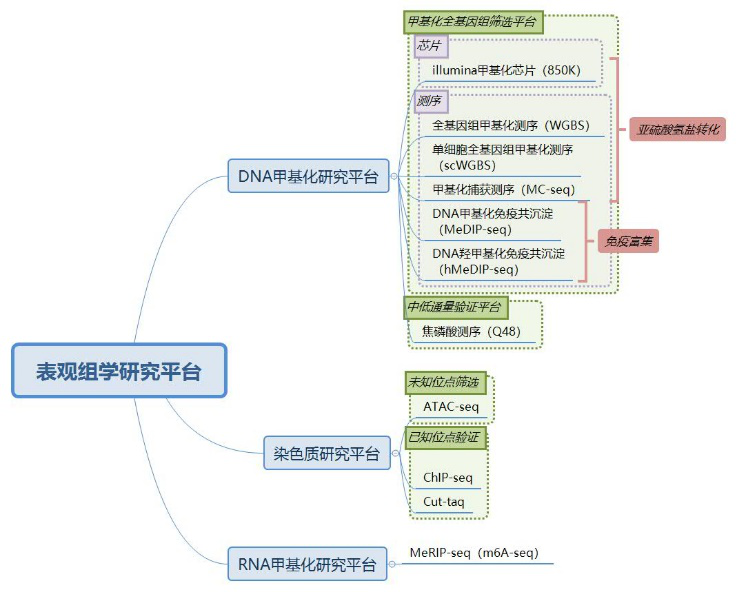
伯豪生物甲基化研究平臺:
更多伯豪生物人工服務(wù):


