信息時代就是好,沒有自己的數據咱可以從免費的數據庫找數據呀!目前 TCGA 和 GEO 數據庫簡直就是專為科研人員準備的免費的寶庫。前面一直講的空間轉錄組,今天來穿插著講一節 TCGA & GEO 數據分析換換口味吧。
今天主要給大家介紹一下對轉錄組 TCGA & GEO 的數據進行分析,包括差異分析、富集分析、單基因展示以及其它個性化做圖。除了想利用公共數據庫挖掘轉錄組數據的發文章的同學之外,這篇教程其實特別適合自己做了少量轉錄組樣本又不想做后期驗證,想直接用公共數據庫的數據來驗證自己數據結果的朋友。
目前各種 TCGA & GEO 數據下載的教程太多了,所以今天直接省略這部分部分,直接從后面的分析開始。廢話不多說,直接上干貨!
一、準備文件
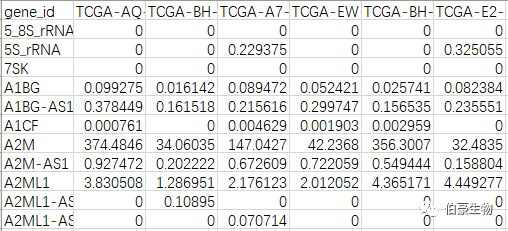
基因表達矩陣文件,可以是 TCGA 下載的轉錄組數據,也可以是 GEO 下載的轉錄組數據,格式如下(行是基因,列是樣本):
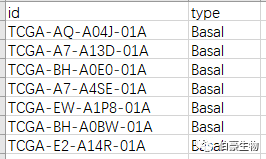
樣本分組文件,格式如下(type 是分組):
二、差異分析
一般 TCGA 或 GEO 上大量樣本數據的差異分析 limma 用的比較多,所以這里也使用 limma 包來分析差異基因,注意需要先安裝 limma R 包。
安裝 limma 包:
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("limma")
分析差異:
## 讀取基因表達件,取 log2
geneexp = read.table("gene_exp_BRCA.txt",header=T,row.names=1,sep="\t")
geneexp = log2(geneexp+1)
## 讀取分組文件
group_file = read.table("sample_group_BRCA.txt",header=T,row.names=1,sep="\t",as.is =TRUE)
rownames(group_file) = gsub('-','.',rownames(group_file))
geneexp = geneexp[,rownames(group_file)]
### 差異分組設置
samps<-factor(group_file$type)
design <- model.matrix(~0+samps) ;
colnames(design) <- levels(samps)
### 模型擬合
fit <- lmFit(geneexp, design)
cont.matrix<-makeContrasts(Basal-Normal,levels=design)
fit2 <- contrasts.fit(fit, cont.matrix)
fit2 <- eBayes(fit2)
final<-topTable(fit2, coef=1, number=dim(geneexp)[1], adjust.method="BH")
> head(final)
logFC AveExpr t P.Value adj.P.Val B
SDPR -4.358861 2.679133 -46.10828 8.888505e-122 4.738017e-117 267.4862
TPX2 4.030685 3.807811 45.39822 2.571983e-120 6.854979e-116 264.1584
UBE2C 4.089727 3.270516 43.72542 8.428935e-117 1.497681e-112 256.1483
CDC20 3.971916 3.450304 42.85468 6.270780e-115 8.356598e-111 251.8811
FIGF -3.220704 1.583802 -40.93757 1.053838e-110 1.123497e-106 242.2399
FAXDC2 -2.179867 1.637909 -39.05494 2.084335e-106 1.851758e-102 232.4283
三、繪制差異基因散點圖和火山圖
這一步需要用到 ggplot2 繪圖,沒有安裝的話需要先安裝 ggplot2 包。
# 繪制差異基因散點圖和火山圖
library(ggplot2)
g1 = "Normal"
g2 = "Basal"
g1_exp = geneexp[rownames(final),rownames(group_file)[which(group_file$type==g1)]]
g2_exp = geneexp[rownames(final),rownames(group_file)[which(group_file$type==g2)]]
g1_mean = apply(g1_exp,1,mean)
g2_mean = apply(g2_exp,1,mean)
type=rep('No',length(g1_mean))
type[which(final$logFC> 1 & final$adj.P.Val <0.05)] = "Up"
type[which(final$logFC < -1 & final$adj.P.Val < 0.05)] = "Down"
datam = data.frame(g1_mean,g2_mean,logFC=final$logFC,FDR=final$adj.P.Val,type,stringsAsFactors=FALSE)
## 散點圖
ggplot(datam,aes(g1_mean,g2_mean,colour=type))+
geom_point(stat="identity",size=1)+theme(legend.title=element_blank())+scale_color_manual(values =c("Down"='blue',"No"='grey',"Up"='orange'))+
labs(x=paste(g1,'Log2(FPKM+1)'),y=paste(g2,'Log2(FPKM+1)'),title=paste(g2,'VS',g1,sep=""))+
coord_cartesian(ylim=c(0,10),xlim=c(0,10))+geom_segment(aes(x = 0, y = 0, xend = 10, yend = 10),size=1,colour="#999999",linetype="dotted")+theme(plot.title = element_text(hjust = 0.5),title=element_text(face="bold",size=15,colour="black"),axis.title=element_text(face="bold",size=13,colour="black"),axis.text.x=element_text(face="bold",size=12,colour="black"),axis.text.y=element_text(face="bold",size=12,colour="black"),legend.text=element_text(face="bold",size=13,colour="black"))
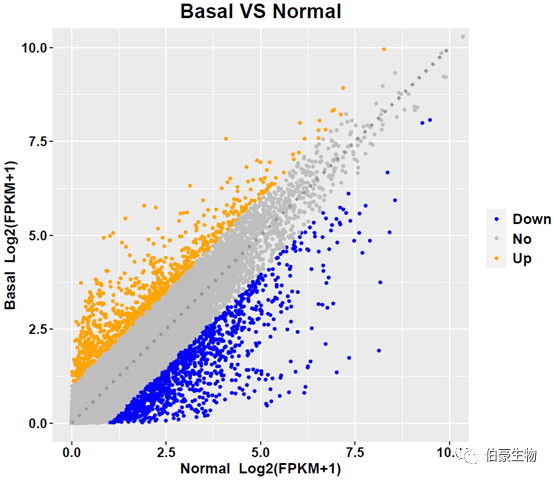
散點圖結果如下:
## 繪制火山圖
ggplot(datam,aes(logFC,-log10(FDR),colour=type))+
geom_point(stat="identity",size=1.2)+theme(legend.title=element_blank())+scale_color_manual(values =c("Down"='blue',"No"='grey',"Up"='orange'))+
labs(x="Log2 (FC)",y="-Lg10 (FDR)",title=paste(g2,'VS',g1,sep=""))+coord_cartesian(xlim=c(-5,5))+
geom_hline(aes(yintercept=1.3),colour="white",size=1.1)+
geom_vline(aes(xintercept =-1),colour="white",size=1.1)+geom_vline(aes(xintercept =1),colour="white",size=1.1)+
theme(axis.title.y = element_text(vjust=-0.1),axis.title.x = element_text(vjust=-0.6),title = element_text(vjust=0.8))+theme(plot.title = element_text(hjust = 0.5),title=element_text(face="bold",size=15,colour="black"),axis.title=element_text(face="bold",size=13,colour="black"),axis.text.x=element_text(face="bold",size=10,colour="black"),axis.text.y=element_text(face="bold",size=10,colour="black"),legend.text=element_text(face="bold",size=12,colour="black"))
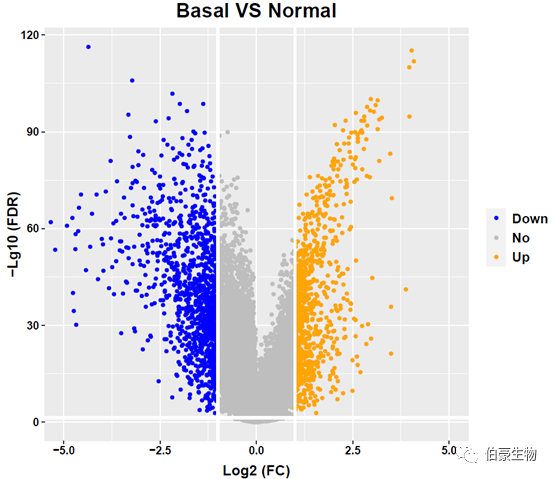
火山圖結果如下:
四、差異基因富集分析
這里我們介紹一下怎么用 clusterProfiler R 包來做差異基因富集分析。需要先安裝好 clusterProfiler,org.Hs.eg.db 兩個 R 包。
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("clusterProfiler")
BiocManager::install("org.Hs.eg.db")
GO 富集分析
library(clusterProfiler)
library(org.Hs.eg.db)
### 提取差異基因 list
diffgenes <- final[(final[,"adj.P.Val"]<0.05 & abs(final[,"logFC"])>=1),]
genelist <- diffgenes[,"logFC"]
names(genelist) = rownames(diffgenes)
##id 轉換,將 SYMBOL 轉換成 ENTREZID
gene = bitr(rownames(diffgenes), fromType = "SYMBOL", toType = "ENTREZID", OrgDb = "org.Hs.eg.db")
#####################GO 富集
ego <- enrichGO(
gene = gene$ENTREZID,
keyType = "ENTREZID",
OrgDb = "org.Hs.eg.db",
ont = "ALL",
pAdjustMethod = "BH",
pvalueCutoff = 0.05,
qvalueCutoff = 0.05,
readable = TRUE)
# 再用 setReadable 函數將基因 ID 映射到基因 Symbol
ego2 <- setReadable(ego, OrgDb = "org.Hs.eg.db", 'ENTREZID')
繪制柱狀圖:
barplot(ego2,showCategory = 20)
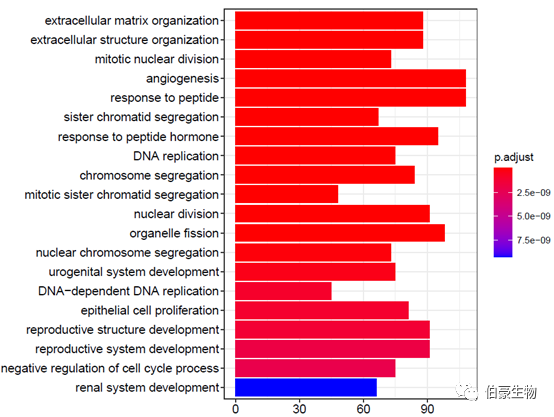
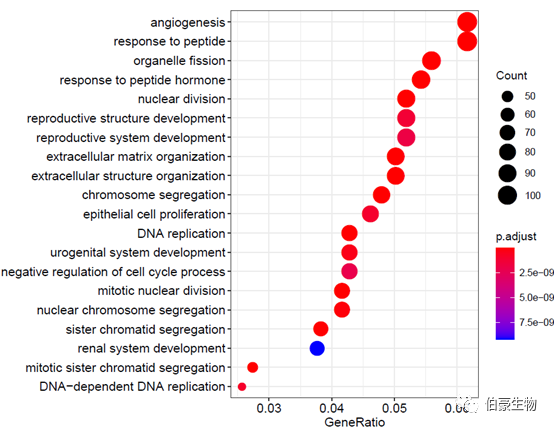
繪制氣泡圖:
dotplot(ego2, showCategory = 20)
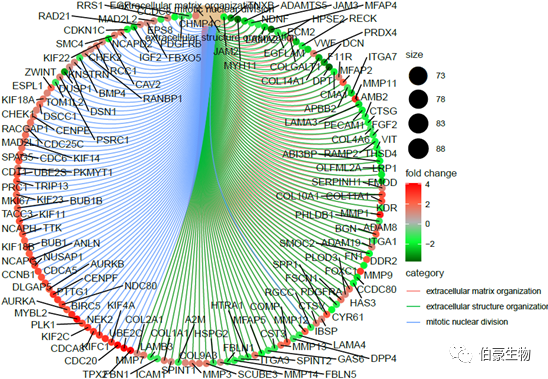
繪制網絡圖 1:
cnetplot(ego2, showCategory = 3,foldChange=genelist, circular = TRUE, colorEdge = TRUE)
繪制網絡圖 2:
emapplot(ego2, foldChange=genelist, showCategory = 20)

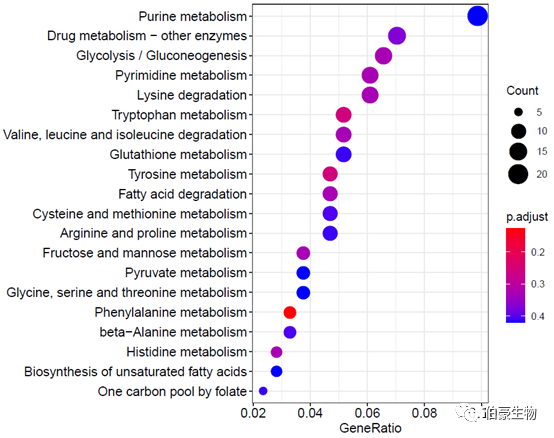
KEGG 富集分析
注意這一步需要連接網絡,因為 clusterProfiler 是在線抓取新的 pathway 數據庫的。當然也有用 kegg.db R 包直接內置數據庫的情況,這里不做介紹。
######################KEGG 富集
kegg <- enrichKEGG(
gene = gene$ENTREZID,
keyType = "kegg",
organism = 'hsa',
pvalueCutoff = 1,
pAdjustMethod = "BH",
qvalueCutoff = 1,
use_internal_data = FALSE)
kegg2 <- setReadable(kegg, OrgDb = "org.Hs.eg.db", 'ENTREZID')
繪制柱狀圖:
barplot(kegg2,showCategory = 20)
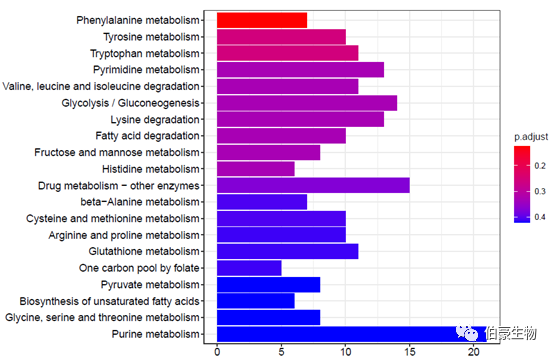
繪制氣泡圖:
dotplot(kegg2, showCategory = 20)
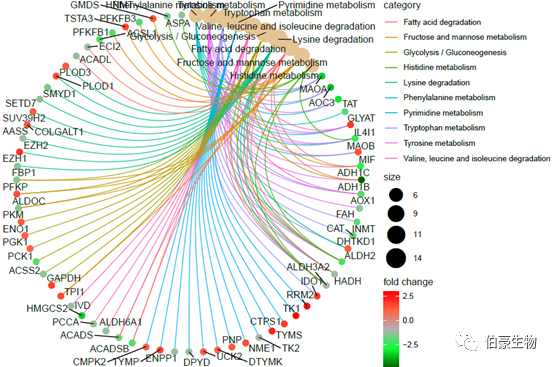
繪制網絡圖 1:
cnetplot(kegg2, showCategory = 10,foldChange=genelist, circular = TRUE, colorEdge = TRUE)
繪制網絡圖 2:
emapplot(kegg2, foldChange=genelist, showCategory = 20)

五、差異基因熱圖標記基因
當我們已經用自己的數據做過分析得到了差異基因,或者是已經有自己關注的基因,想用 TCGA & GEO 數據來驗證自己的數據或結論的時候,用 TCGA & GEO 數據差異基因熱圖同時標記特定基因來展示結果就特別合適。
###### 差異基因熱圖標記關注基因
library("ComplexHeatmap")
library("circlize")
diff_exp = geneexp[rownames(diffgenes),]
# 進行 zscore 歸一化
diff_exp_scaled = t(apply(diff_exp, 1, scale))
colnames(diff_exp_scaled) = colnames(diff_exp)
# 需要標記的基因
genes = c('CDC20','THRB','FAM13A','CCNA2','PRR15','CHI3L1','CLCA2','ABCA10','A2ML1','LCN2')
## 設置位置
gene_pos = which(rownames(diff_exp) %in% genes)
ha = rowAnnotation(foo = anno_mark(at = gene_pos, labels = genes))
m=round(max(abs(diff_exp_scaled)))
## 畫熱圖
Heatmap(diff_exp_scaled, colorRamp2(c(-m,0,m),c("blue", "#EEEEEE", "red")),
right_annotation = ha,name = "Z-score",show_row_names =FALSE,show_column_names =FALSE)
繪圖結果如下:

六、單個基因繪圖
要在 TCGA & GEO 數據中驗證自己的關注的基因的差異情況,除了前面說的差異基因熱圖標記特定基因之外,也可以對單個基因直接進行繪圖。這里我們提供三種展示方式,選擇自己喜歡的一種展示形式就行。
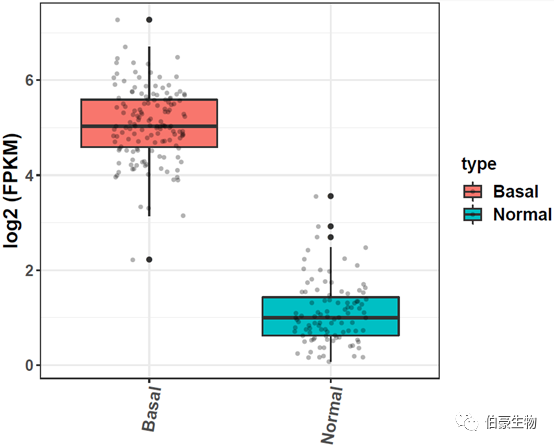
用箱線圖展示:
## 單個基因箱線圖
exp2 = data.frame(t(diff_exp))
exp2$type = group_file$type
ggplot(exp2, aes(x=type, y=CDC20,fill=type)) +
geom_boxplot()+geom_jitter(alpha = .3, width =0.2,size=1)+labs(title = "") + ylab("log2 (FPKM)")+ xlab("")+theme_bw()+
theme(title=element_text(face="bold",size=16),axis.title=element_text(face="bold",size=15),axis.text.x=element_text(face="bold",angle=80,size=13,hjust=1),
axis.text.y=element_text(face="bold",size=12),legend.text=element_text(face="bold",size=13),legend.title=element_text(face="bold",size=13))
結果如下:
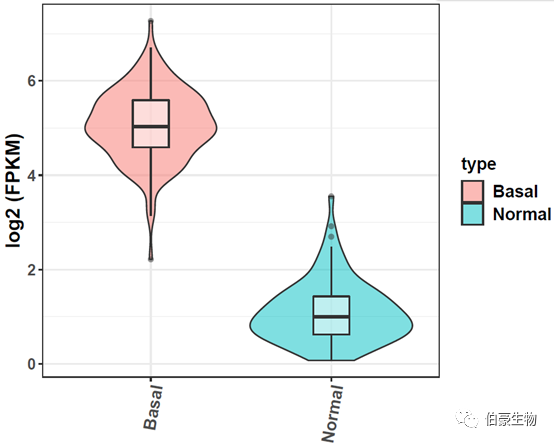
小提琴圖展示:
### 單個基因小提琴圖
ggplot(exp2, aes(x=type, y=CDC20,fill=type)) +
geom_violin(alpha=0.5) + geom_boxplot(alpha = .5,fill="white", width= .2)+labs(title = "") + ylab("log2 (FPKM)")+ xlab("")+theme_bw()+
theme(title=element_text(face="bold",size=16),axis.title=element_text(face="bold",size=15),axis.text.x=element_text(face="bold",angle=80,size=13,hjust=1),
axis.text.y=element_text(face="bold",size=12),legend.text=element_text(face="bold",size=13),legend.title=element_text(face="bold",size=13))
結果如下:
散點圖展示:
### 單個基因散點圖
ggplot(exp2, aes(x=type, y=CDC20)) +
geom_dotplot(binaxis='y', stackdir='center',binwidth = 0.1)+stat_summary(fun.y=median, geom="point", shape=18,size=3, color="red")+
theme(axis.title=element_text(face="bold",size=15),axis.text.x=element_text(face="bold",angle=80,size=13,hjust=1),axis.text.y=element_text(face="bold",size=12))
結果如下:
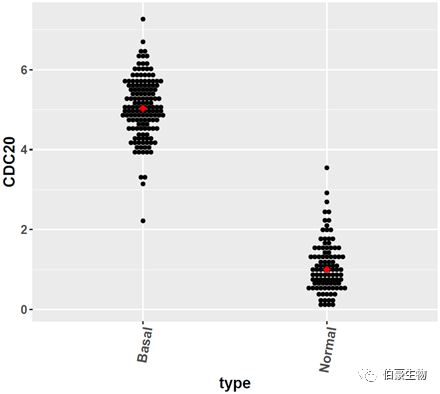
這里只展示一個基因的情況,如果是有多個基因需要繪圖的,把代碼里的基因名替換一下就好了。
好啦,今天的分享就到次為止啦,下次再繼續哦!
更多伯豪生物人工服務:


