單細(xì)胞 RNA 測(cè)序(scRNA-seq)技術(shù)的飛速發(fā)展使人們對(duì)組織中細(xì)胞種類、細(xì)胞的特殊狀態(tài)有了深入認(rèn)識(shí)。 但是,scRNA-seq 對(duì)于器官或者固體組織制備的細(xì)胞懸液的細(xì)胞活性和細(xì)胞數(shù)目有著較高的要求,這也意味著“躺在”超低溫冰箱中的大量珍貴臨床樣本(腦組織,腫瘤組織等)都無法進(jìn)行 scRNA 測(cè)序。 而單細(xì)胞核 RNA 測(cè)序技術(shù)(snRNA-seq)的出現(xiàn),則在很大程度上解決了以上問題。
雖然細(xì)胞核內(nèi)的遺傳物質(zhì)可以大體代表整個(gè)細(xì)胞,然而,細(xì)胞質(zhì)和細(xì)胞核之間的 RNA 類型和比例卻存在一定的差異。RNA 首先在細(xì)胞核內(nèi)轉(zhuǎn)錄,并在細(xì)胞核內(nèi)積累到穩(wěn)定狀態(tài)。在細(xì)胞質(zhì)中,mRNA 根據(jù)其出核率和不同的命運(yùn),包括轉(zhuǎn)運(yùn)到特定的亞細(xì)胞位置、核糖體的翻譯、小 RNA 的降解等,達(dá)到不同的穩(wěn)態(tài)水平。研究人員發(fā)現(xiàn),細(xì)胞核 RNA 中含有大量的非編碼序列,其中包含 41% 的基因間區(qū)序列和 25% 的內(nèi)含子序列。同時(shí),研究人員也發(fā)現(xiàn)有 41.7% 的轉(zhuǎn)錄本序列只在細(xì)胞核中存在。多項(xiàng)研究表明,將內(nèi)含子區(qū)域的 reads 增加了 snRNA-seq 的敏感性,并提高識(shí)別細(xì)胞類型的分辨率。因此,在處理單細(xì)胞核 RNA 測(cè)序數(shù)據(jù)時(shí),納入內(nèi)含子序列具有重要意義。下面,就讓我們?cè)敿?xì)了解一下針對(duì)不同組織樣本進(jìn)行 snRNA-seq 的部分案例與要點(diǎn)。
腦組織樣本的 snRNA-seq
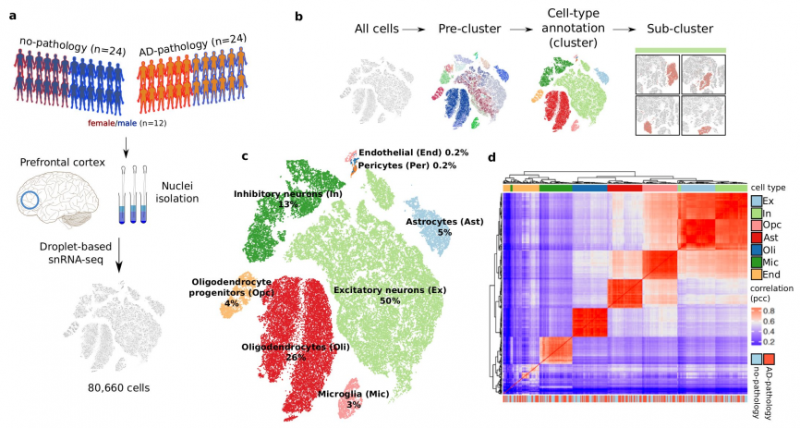
對(duì)于腦組織來說,scRNA-seq 并不能全面地分析神經(jīng)細(xì)胞的類型。一些細(xì)胞類型更容易受到組織解離過程的影響,比如在成人的新大腦皮層中,非神經(jīng)元細(xì)胞在解離中比神經(jīng)元耐受性更好,在單細(xì)胞懸浮液中大量存在;在小鼠新皮層中,第 5 層 parvalbuin 陽性的間神經(jīng)元和皮質(zhì)下突起的谷氨酸能神經(jīng)元的比例低于預(yù)期。相反,細(xì)胞核對(duì)機(jī)械處理更有抵抗力,可以從冷凍組織中解離出來。 研究發(fā)現(xiàn),單個(gè)細(xì)胞核已被證明可以提供足夠的基因表達(dá)信息來定義成人大腦和小鼠海馬區(qū)相對(duì)寬泛的細(xì)胞類別。 此外,新鮮解離的神經(jīng)組織很難獲得;相比之下,從意外去世或者因疾病去世后病人處所獲得的正常和疾病組織,是研究大腦組織中細(xì)胞類型的主要樣本類型。 但是,這些組織通常是冰凍的樣本,因此 snRNA-seq 是解決細(xì)胞類型少以及冰凍樣本的重要方法。
研究發(fā)現(xiàn),從冷凍的 S1 皮層中分離單個(gè)核,進(jìn)行神經(jīng)核抗原(NeuN) 流動(dòng)分選,并在 Fluidigm C1 系統(tǒng)上進(jìn)行 RNA 測(cè)序,細(xì)胞核和細(xì)胞數(shù)據(jù)顯示檢測(cè)到的基因數(shù)量和類型相似(S1 細(xì)胞核 - 平均 5619 個(gè)基因;S1 細(xì)胞 - 平均 4797 個(gè)基因)。核數(shù)核據(jù)含有較高比例的內(nèi)含子 reads。
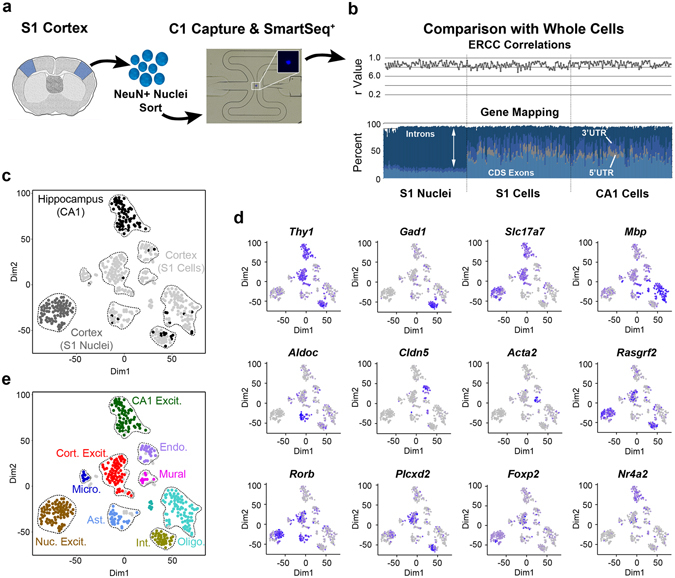
圖。SnRNA-seq 揭示了興奮性神經(jīng)元的特性
Grubman?等人通過對(duì)從去世后的阿爾茨海默氏癥患者和年齡匹配的非疾病患者身上提取的內(nèi)嗅皮層組織進(jìn)行 snRNA-seq,得到了 13,214 個(gè)細(xì)胞,每個(gè)細(xì)胞中位數(shù)檢測(cè)到 646 個(gè)基因。UMAP 顯示,共鑒定到了小膠質(zhì)細(xì)胞、星形膠質(zhì)細(xì)胞、神經(jīng)元、少突膠質(zhì)祖細(xì)胞(OPCs),少突膠質(zhì)細(xì)胞和內(nèi)皮細(xì)胞。
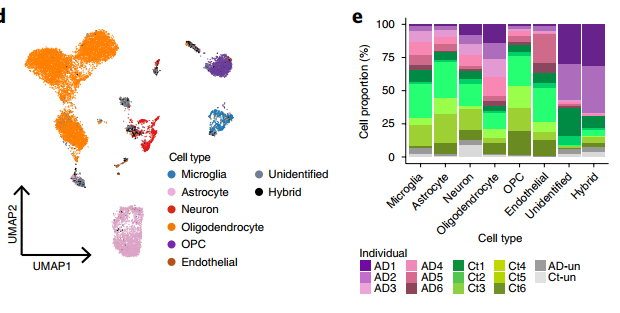
圖。人內(nèi)嗅皮層 snRNA-seq 揭示阿爾茨海默病中細(xì)胞類型特異性標(biāo)記基因和細(xì)胞類型特異性改變
同樣是阿爾茲海默癥疾病,Mathys 等人通過對(duì)冷凍的去世后病人腦組織進(jìn)行 snRNA-seq,共鑒定到了人類大腦的主要細(xì)胞類型:興奮性神經(jīng)元(NRGN 標(biāo)記),抑制性神經(jīng)元(GAD1),星形膠質(zhì)細(xì)胞(AQP4),少突膠質(zhì)細(xì)胞(MBP),小膠質(zhì)細(xì)胞(CSF1R 和 CD74)、少突膠質(zhì)祖細(xì)胞(VCAN)、內(nèi)皮細(xì)胞和周細(xì)胞(AMBP)。
圖。snRNA-seq 譜分析阿爾茲海默癥病人腦組織中細(xì)胞類型
心臟組織樣本的 snRNA-seq
在心臟的單細(xì)胞 RNA 測(cè)序研究中也存在著許多難點(diǎn)。 首先,成年的心臟組織較難消化,很難保證在不損傷細(xì)胞的情況下,得到高質(zhì)量的細(xì)胞懸液;再者,由于心肌細(xì)胞大,且形狀不規(guī)則,導(dǎo)致細(xì)胞捕獲較難。因此,心臟的研究通常依賴胚胎和新生兒的小鼠心臟或關(guān)注成年小鼠心臟的非心肌細(xì)胞群來避免這些問題。2020 年發(fā)表的一篇關(guān)于哺乳動(dòng)物心臟單細(xì)胞核 RNA 測(cè)序探索細(xì)胞成分和細(xì)胞特征的文章,則在一定程度上掃除了心臟單細(xì)胞 RNA 測(cè)序的障礙。snRNA-seq 分析得到了 8635 個(gè)細(xì)胞核和 22,568 個(gè)基因,每個(gè)細(xì)胞平均得到 2662.6 reads。UMAP 顯示了 24 個(gè)不同的細(xì)胞類群,其中較多的是內(nèi)皮細(xì)胞(28.8%)、成纖維細(xì)胞(25.3%) 和心肌細(xì)胞(22.8%),它們分別含有約 2500 個(gè)、2200 個(gè)和 2000 個(gè)細(xì)胞核。
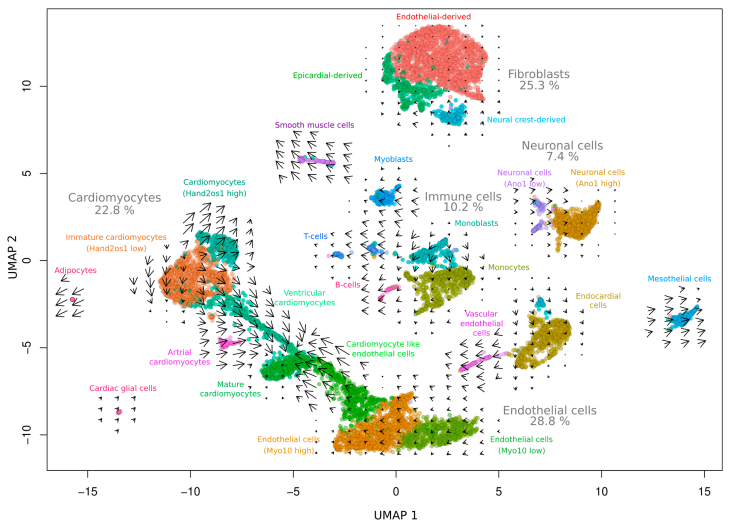
圖。snRNA-seq 譜分析心臟組織中細(xì)胞類型
Cui 等人使用 snRNA-seq 揭示再生新生兒和非再生晚期心臟心肌細(xì)胞的不同群體。結(jié)果表明,一個(gè)明顯的未成熟的心肌細(xì)胞群體進(jìn)入細(xì)胞周期響應(yīng)損傷。對(duì)這些心肌細(xì)胞的轉(zhuǎn)錄組分析揭示了支持新生兒再生反應(yīng)的基因調(diào)控網(wǎng)絡(luò),該網(wǎng)絡(luò)的缺失與再生阻斷相一致。
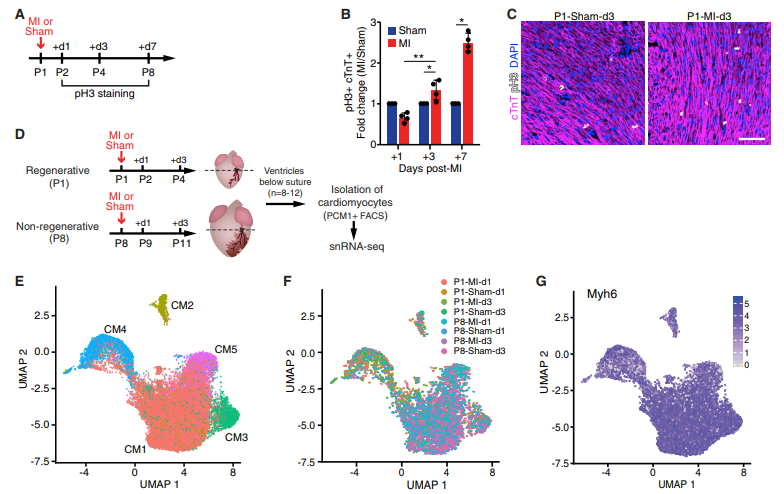
圖。snRNA-seq 確定新生兒心臟中不同的心肌細(xì)胞群
腎臟組織樣本的 snRNA-seq
2019 年,來自華盛頓大學(xué)醫(yī)學(xué)院的 Benjamin D. Humphreys 團(tuán)隊(duì)比較分析了 腎臟組織的單細(xì)胞 RNA 測(cè)序(scRNA-seq) 和腎臟組織的單細(xì)胞核 RNA 測(cè)序(snRNA-seq) 在腎臟細(xì)胞類群鑒定中的區(qū)別。 與 scRNA-seq 相比,snRNA-seq 捕獲了多種在 scRNA-seq 數(shù)據(jù)集中不存在的腎臟細(xì)胞類型,包括腎小球足細(xì)胞、系膜細(xì)胞和內(nèi)皮細(xì)胞。同時(shí)也未檢測(cè)到應(yīng)激反應(yīng)基因。與已發(fā)表的 scRNA-seq 數(shù)據(jù)集(分別為 2.4% 和 0.12%) 相比,snRNA-seq 方法產(chǎn)生了 20 倍以上的足細(xì)胞。
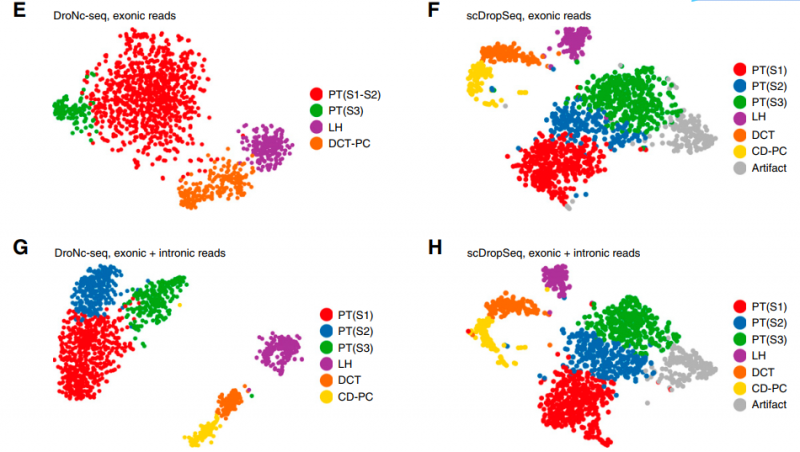
圖。加上內(nèi)含子 reads,snRNA 測(cè)序的表現(xiàn)相當(dāng)于或優(yōu)于 scRNA 測(cè)序
Wilson 等人通過對(duì)冷凍保存的糖尿病人腎臟樣本進(jìn)行 snRNA-seq,共得到 23,980 單細(xì)胞核數(shù)據(jù),其中包括 11 種腎臟細(xì)胞類型和 4 種免疫細(xì)胞類型(近曲小管;CFH,補(bǔ)體因子 H 陽性細(xì)胞;亨利循環(huán)細(xì)胞;遠(yuǎn)曲小管細(xì)胞;連接小管細(xì)胞;集尿管細(xì)胞;主細(xì)胞;插入細(xì)胞;阿足突細(xì)胞;內(nèi)皮細(xì)胞;系膜細(xì)胞;白細(xì)胞)。
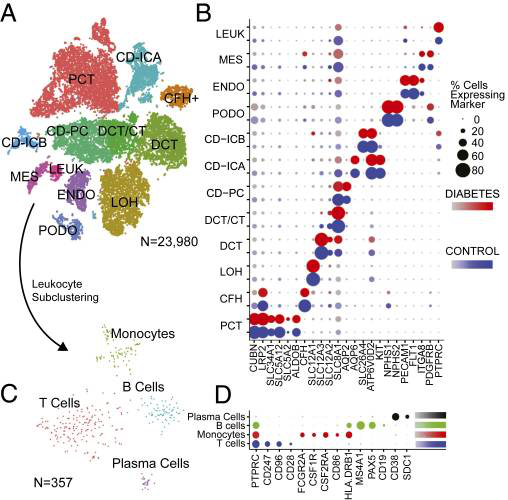
圖。snRNA 測(cè)序鑒定腎臟細(xì)胞及免疫細(xì)胞種類
結(jié) 語
總結(jié)而言,單細(xì)胞核測(cè)序技術(shù)在樣本適用性上較單細(xì)胞 RNA 測(cè)序技術(shù)更有優(yōu)勢(shì),它不局限于新鮮的組織樣本,適用于冰凍樣本。此外,單細(xì)胞核的制備相對(duì)于單細(xì)胞懸液的制備要更簡(jiǎn)單,可以盡量減少酶解,機(jī)械壓力誘導(dǎo)的假的細(xì)胞類群的產(chǎn)生。在數(shù)據(jù)分析方面,單細(xì)胞核 RNA 測(cè)序可以獲得內(nèi)含子區(qū)、基因間區(qū)的數(shù)據(jù),使細(xì)胞類型的鑒定分辨率更高,獲得的基因信息相對(duì)更加豐富。
但是單細(xì)胞核測(cè)序可以取代單細(xì)胞 RNA 測(cè)序么? 答案一定是 NO! 在選擇兩種方法時(shí),要更多的結(jié)合自身的條件來確定。自己有什么樣本?是否可以制備合格的細(xì)胞懸液?是否有對(duì)酶敏感的細(xì)胞類型?各項(xiàng)因素都需要綜合考慮。未來,單細(xì)胞核 RNA 測(cè)序與單細(xì)胞 RNA 測(cè)序的結(jié)合可能是全面獲得樣本中細(xì)胞類型的策略。也讓我們期待廣大的科研工作者來解密。
伯豪生物單細(xì)胞多組學(xué)研究一站式服務(wù)優(yōu)勢(shì)
平臺(tái)齊全
擁有 BD、10X Genomics、SMART-seq 三大單細(xì)胞測(cè)序服務(wù)平臺(tái)。
經(jīng)驗(yàn)豐富
累計(jì)制備 50 余種組織類型的單細(xì)胞樣本,細(xì)胞活率平均 90% 以上。
樣本制備
單細(xì)胞測(cè)序樣本保存液、單細(xì)胞核分離試劑盒以及單細(xì)胞懸液制備方法完美解決單細(xì)胞樣本制備問題。
數(shù)據(jù)分析
建立了完善的單細(xì)胞測(cè)序(核)數(shù)據(jù)處理方法及個(gè)性化分析方法和流程。
參考文獻(xiàn)
1. Wilson P C, Wu H, Kirita Y, et al. The single-cell transcriptomic landscape of early human diabetic nephropathy[J]. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(39): 19619-19625.
2. Lake B B, Ai R, Kaeser G E, et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain[J]. Science, 2016, 352(6293): 1586-1590.
3. Bakken T E, Hodge R D, Miller J A, et al. Single-nucleus and single-cell transcriptomes compared in matched cortical cell types[J]. PLOS ONE, 2018, 13(12).
4. Selewa A, Dohn R, Eckart H, et al. Systematic Comparison of High-throughput Single-Cell and Single-Nucleus Transcriptomes during Cardiomyocyte Differentiation.[J]. Scientific Reports, 2020, 10(1).
5. Lake B B, Codeluppi S, Yung Y C, et al. A comparative strategy for single-nucleus and single-cell transcriptomes confirms accuracy in predicted cell-type expression from nuclear RNA[J]. Scientific Reports, 2017, 7(1): 6031-6031.
6. Barthelson R, Lambert G M, Vanier C H, et al. Comparison of the contributions of the nuclear and cytoplasmic compartments to global gene expression in human cells[J]. BMC Genomics, 2007, 8(1): 340-340.
7. Zhang Y, Gagolopez N, Li N, et al. Single-cell imaging and transcriptomic analyses of endogenous cardiomyocyte dedifferentiation and cycling.[J]. Cell discovery, 2019, 5(1).
8. MiaoCui,ZhaoningWang,KenianChen,AkanshaM.et al. Dynamic Transcriptional Responses to Injury of Regenerative and Non-regenerative Cardiomyocytes Revealed by Single-Nucleus RNA Sequencing[J]. Dev Cell . 2020 Apr 6;53(1):102-116.e8.
9. Grubman A, Chew G, Ouyang J F, et al. A single-cell atlas of entorhinal cortex from individuals with Alzheimer’s disease reveals cell-type-specific gene expression regulation[J]. Nature Neuroscience, 2019, 22(12): 2087-2097.
10. Wolfien M, Galow A, Muller P, et al. Single-Nucleus Sequencing of an Entire Mammalian Heart: Cell Type Composition and Velocity[J]. Cells, 2020, 9(2).
11. Mathys H, Davilavelderrain J, Peng Z, et al. Single-cell transcriptomic analysis of Alzheimer’s disease[J]. Nature, 2019, 570(7761): 332-337.
12. Wu H, Kirita Y, Donnelly E L, et al. Advantages of Single-Nucleus over Single-Cell RNA Sequencing of Adult Kidney: Rare Cell Types and Novel Cell States Revealed in Fibrosis[J]. Journal of The American Society of Nephrology, 2019, 30(1): 23-32.
更多伯豪生物人工服務(wù):


