為什么要選擇單細胞核 RNA 測序而非單細胞 RNA 測序?
單細胞核 RNA 測序的優勢:
單細胞 RNA 測序技術的高通量和敏感性使其非常適合于全面繪制細胞狀態的變化。但是在腎臟和其他固體組織中(尤其是對于高上皮含量的組織,以及以細胞外基質為特征的固體組織),單細胞 RNA 測序研究的一個大的挑戰是如何獲得高質量的單細胞懸浮液,高質量的單細胞懸浮液應該包含罕見或難以解離的細胞類型,細胞的 mRNA 不降解,基因的表達不受解離反應的影響。但是現實實驗得到的結果確不是這樣的,相信做過單細胞測序的科研人員都會遇到以下這樣的問題:
單細胞 RNA 測序不能準確地捕獲組織中所有細胞類型(比如腎臟,腦),因為單細胞解離本身可能損害敏感細胞。
目前用于單細胞解離的酶和機械方法會引入了因裂解壓力誘導的轉錄產物。
活性蛋白酶傾向于選擇易解離的細胞類型。不易解離的細胞可能會被丟失。
目前的方法與冷凍樣本材料不兼容(冷凍樣本不能用于單細胞測序),但是有的時候,臨床所取的樣本需要等待病理診斷后才能去做單細胞測序(比如腎臟活檢樣本),因此為了保證樣本的 RNA 穩定性,需要進行凍存。
基于以上 4 點局限性:
2019 年,來自華盛頓大學醫學院的 Benjamin D. Humphreys 團隊通過比較分析了腎臟組織的單細胞 RNA 測序(scRNA-seq) 和腎臟組織的單細胞核 RNA 測序(snRNA-seq),在腎臟細胞類群鑒定中的區別,該研究成果發表在腎臟病學頂尖國際期刊 J Am Soc Nephrol 上(Advantages of Single-Nucleus over Single-Cell RNA Sequencing of Adult Kidney: Rare Cell Types and Novel Cell States Revealed in Fibrosis)。
研究人員將使用 DropSeq 平臺的 (scRNA-seq 與使用 snuco -DropSeq、DroNc-seq 和 10X genomics 三個平臺的 snRNA-seq 結果進行了比較。并驗證了 snRNA-seq 對小鼠單側輸尿管梗阻(UUO) 術后 14 天腎臟纖維化的作用。
scRNA-seq 測序結果顯示,腎臟組織中共獲得了 10 個細胞類群,但腎小球細胞類型缺失,此外,其中一個細胞類群主要由人為解離誘導的應激反應基因組成。相比之下,snRNA-seq 捕獲了多種在 scRNA-seq 數據集中不存在的腎臟細胞類型,包括腎小球足細胞、系膜細胞和內皮細胞。同時也未檢測到應激反應基因。與已發表的 scRNA-seq 數據集(分別為 2.4% 和 0.12%) 相比,snRNA-seq 方法產生了 20 倍以上的足細胞。令人意外的是,scRNA-seq 平臺和 snRNA-seq 平臺的基因檢測靈敏度相當。為了驗證這一結論,對冷凍的第 14 天 UUO 腎臟的分析顯示了罕見的腎小球旁細胞、新激活的近端小管和成纖維細胞狀態,以及以前未識別的小管間質信號通路。
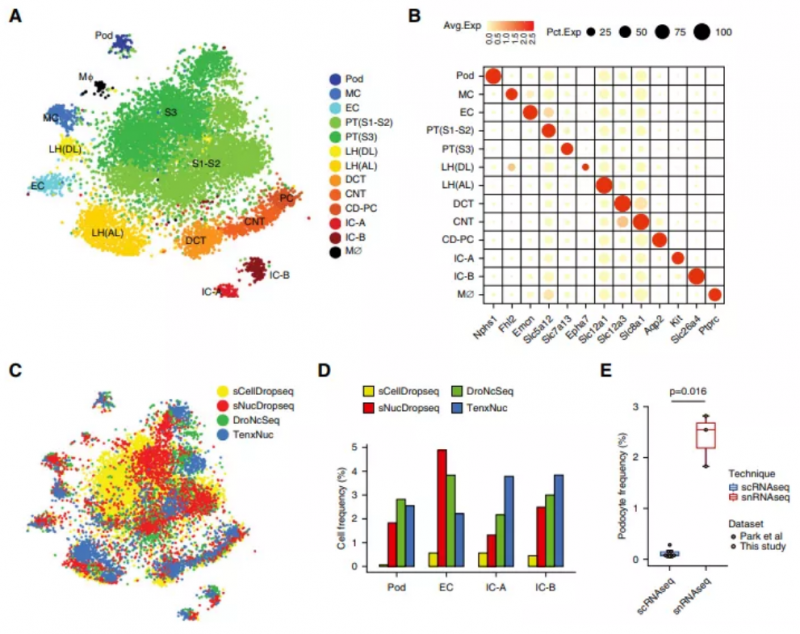
圖:snRNA-seq 相對于 scRNA-seq 技術具有較低的解離偏差。(A) tSNE 顯示了 13 個細胞類群。(B) 不同類群的標記基因表達。(C) tSNE 顯示每個平臺的數據對所有集群的貢獻。(D) 每個平臺貢獻的細胞百分比顯示,與 snRNA-seq 相比,scRNA-seq 技術對足細胞、內皮細胞和夾層細胞的檢出率非常低 (E) snRNA-seq(2.4%)檢測出的足細胞是 scRNA-seq(0.12%)的 20 倍。
綜上所述,與 scRNAseq 相比,snRNA-seq 提供了更少的細胞解離偏倚和等效的基因檢測。盡管 snRNA-seq 在不同基因中所占比例(7%) 低于 scRNA-seq,但其中許多是線粒體或人為應激反應基因,細胞鑒定沒有受到損害。
此外,我們也整理了近年來針對于腦組織的單細胞測序的文章,發現針對于腦組織的單細胞測序, 通常選擇的是 snRNA-seq 而非 scRNA-seq,例如:




由此可見,snRNA-seq 相比于 scRNA-seq 在高上皮含量的組織,以及以細胞外基質為特征的固體組織的單細胞 RNA 測序方面,可以有效地將組織還原為單細胞核分離物,并獲得高精度的細胞類型表達譜。
微信原文:https://mp.weixin.qq.com/s/j5-p7jAC4UWh2H5car_y7Q
更多伯豪生物服務人工服務


